[摘要] 背景与目的:DNA异常甲基化与肿瘤的发生、发展密切相关。本研究旨在探索胰腺癌(pancreatic adenocarcinoma,PAAD)甲基化驱动基因(methylation-driven gene,MDG)中间丝家族孤儿蛋白1(intermediate filament family orphan 1,IFFO1)在PAAD中的表达、对PAAD细胞侵袭转移的影响,以及作为诊断和预后标志物的潜力。方法:从癌症基因组图谱(The Cancer Genome Atlas,TCGA)数据库、国际癌症基因组联盟(International Cancer Genome Consortium,ICGC)数据库、高通量基因表达(Gene Expression Omnibus,GEO)数据库获取PAAD及其癌旁组织的mRNA表达数据(TCGA-PAAD-mRNA)、DNA甲基化芯片数据(TCGA-PAAD-meth、GSE53051、PACA-AU)以及健康人血液的DNA甲基化芯片数据(GSE69270)。通过差异表达分析联合差异甲基化分析筛选PAAD的MDG。在TCGA数据库中,应用Pearson相关性检验验证IFFO1启动子甲基化水平与其表达水平之间的相关性。同时,通过Kaplan-Meier生存分析评估IFFO1启动子甲基化水平、表达水平与PAAD预后的关系。27例PAAD患者的癌组织及其对应癌旁组织的病理切片来自上海交通大学医学院附属第一人民医院。本研究涉及的所有样本均通过上海交通大学医学院附属第一人民医院人类伦理委员会的审核批准(伦理编号:院伦审[2017]53号)。利用免疫组织化学染色(immunohistochemistry staining,IHC)检测27例PAAD患者的癌组织及其对应癌旁组织中IFFO1的表达。根据IFFO1表达的中位值,将TCGA数据库中的患者分为高表达组和低表达组,并进行差异分析、基因本体论(gene ontology,GO)富集分析及基因集富集分析(gene set enrichment analysis,GSEA)。使用蛋白质印迹法(Western blot)及实时荧光定量聚合酶链反应(real-time fluorescence quantitative polymerase chain reaction,RTFQ-PCR)检验正常胰腺导管上皮细胞系H6C7与PAAD细胞系MIA PaCa2、BxPC-3、AsPC-1和Capan-2中IFFO1的表达差异。通过细胞划痕实验和侵袭实验检测过表达IFFO1对PAAD细胞AsPC-1和Capan-2的迁移和侵袭能力的影响;通过受试者工作特征(receiver operating characteristic,ROC)曲线和Kaplan-Meier生存分析评估IFFO1甲基化水平在TCGA泛癌队列中的诊断和预后预测价值。结果:通过对5组数据的交叉筛选,筛选出41个PAAD的MDG。其中,IFFO1与PAAD的预后关系最为密切[风险比(hazard ratio,HR)=0.28,P<0.001]。IFFO1在PAAD中高甲基化而低表达,其启动子的甲基化水平与表达水平呈显著负相关关系(r=-0.55,P<0.001)。IHC结果显示,IFFO1在PAAD组织中的表达显著低于癌旁组织(P<0.05)。TCGA生存分析结果表明,高甲基化或低表达IFFO1的患者总生存期更差(P<0.05)。GO和GSEA富集分析结果均表明,迁移和运动的负向调控通路在IFFO1高表达患者中富集。Western blot和RTFQ-PCR结果显示,IFFO1在正常胰腺导管上皮细胞系H6C7中的表达显著高于PAAD细胞系MIA PaCa2、BxPC-3、AsPC-1和Capan-2。过表达IFFO1可显著抑制PAAD细胞株AsPC-1和Capan-2的迁移和侵袭。进一步泛癌分析结果提示,IFFO1普遍存在启动子高甲基化而低表达的现象,其甲基化水平在多种癌症中均表现出良好的诊断和预后预测价值。结论:启动子高甲基化导致IFFO1在PAAD中低表达,IFFO1能够抑制PAAD细胞的侵袭和迁移能力。IFFO1甲基化有望成为PAAD诊断和预后的新型标志物。
[关键词] 中间丝家族孤儿蛋白1;胰腺癌;侵袭转移;DNA甲基化
[Abstract] Background and purpose: Abnormal DNA methylation is closely associated with the onset and progression of tumors. This study aimed to investigate the expression of intermediate filament family orphan 1 (IFFO1), a methylation-driven gene (MDG) in pancreatic adenocarcinoma (PAAD), along with its effects on the invasion and metastasis of PAAD cells, as well as its potential as a diagnostic and prognostic biomarker. Methods: mRNA expression data (TCGA-PAAD-mRNA), DNA methylation data (TCGA-PAAD-meth, GSE53051, PACA-AU) of PAAD and adjacent normal tissues, as well as DNA methylation data of healthy individuals’ blood (GSE69270), were obtained from the The Cancer Genome Atlas (TCGA), International Cancer Genome Consortium (ICGC) and Gene Expression Omnibus (GEO) databases. By performing differential expression analysis combined with differential methylation analysis, we screened for MDG in PAAD. In the TCGA database, Pearson correlation tests were employed to verify the relationship between IFFO1 promoter methylation level and its expression level. Additionally, Kaplan-Meier survival analysis was conducted to evaluate the relationship among IFFO1 promoter methylation level, expression level, and the prognosis of PAAD. Pathological ps of cancer tissues and corresponding adjacent tissues from 27 PAAD patients were obtained from Shanghai General Hospital, Shanghai Jiao Tong University School of Medicine. All samples involved in this study were approved by the human ethics committee of Shanghai General Hospital, Shanghai Jiao Tong University School of Medicine (ethics number: hospital ethics review[2017]No.53). Immunohistochemistry staining (IHC) was utilized to detect the expression of IFFO1 in cancer tissues and corresponding adjacent tissues from 27 PAAD patients. Based on the median expression level of IFFO1, patients in the TCGA database were classified into high-expression and low-expression groups. Subsequently, differential analysis, gene ontology (GO) enrichment analysis and gene set enrichment analysis (GSEA) were performed. Western blot and real-time fluorescence quantitative polymerase chain reaction (RTFQ-PCR) were employed to assess the expression variations of IFFO1 between the normal pancreatic ductal epithelial cell line H6C7 and the PAAD cell lines MIA PaCa2, BxPC-3, AsPC-1 and Capan-2. The impact of IFFO1 overexpression on the migration and invasion capacities of PAAD cell lines AsPC-1 and Capan-2 was evaluated using scratch and invasion assays. Additionally, receiver operating characteristic (ROC) curves and Kaplan-Meier survival analysis were utilized to assess the diagnostic and prognostic significance of IFFO1 methylation levels in the TCGA pan-cancer cohort. Results: Through the cross-screening of five datasets, 41 MDG in PAAD were identified. Among these, IFFO1 was found to be the gene most closely associated with the prognosis of PAAD [hazard ratio (HR)=0.28, P<0.001]. IFFO1 exhibited high methylation and low expression levels in PAAD. Moreover, a significant negative correlation was observed between the methylation level of its promoter and its expression level (r=-0.55, P<0.001). IHC results indicated that IFFO1 expression was significantly lower in PAAD tissues than in adjacent non-tumor tissues (P<0.05). TCGA survival analysis demonstrated that patients with high methylation or low expression of IFFO1 had poorer overall survival (P<0.05). Both GO and GSEA analyses indicated that the pathway “Negative regulation of cell migration” was enriched in patients with high IFFO1 expression. Western blot and RTFQ-PCR results demonstrated that IFFO1 expression in normal pancreatic ductal epithelial cells H6C7 was significantly higher compared with PAAD cell lines MIA PaCa2, BxPC-3, AsPC-1, and Capan-2. Overexpression of IFFO1 significantly inhibited the migration and invasion of the PAAD cell lines AsPC-1 and Capan-2. Additionally, pan-cancer analysis revealed that IFFO1 exhibited abnormal promoter methylation and low expression across various cancer types, with its methylation levels demonstrating significant diagnostic and prognostic prediction value among different tumors. Conclusion: Promoter hypermethylation results in decreased expression of IFFO1 in PAAD. IFFO1 may suppress the invasion and migration abilities of PAAD cells. Furthermore, IFFO1 methylation holds great promise as a novel biomarker for the diagnosis and prognosis of PAAD.
[Key words] Intermediate filament family orphan 1; Pancreatic adenocarcinoma; Invasion and migration; DNA methylation
胰腺癌(pancreatic adenocarcinoma,PAAD)是一种高度恶性的消化道肿瘤,也是全球癌症相关死亡的主要原因之一。据统计,2023年美国PAAD死亡病例50 550例,新发病例64 050例,年死亡率排名第3位[1]。中国PAAD的发病率不断增加,患者预后极差,5年生存率不足10%[2-3]。由于PAAD起病症状隐匿,且解剖位置特殊,往往难以早期发现。超过50%的患者就诊时已出现远处转移,而伴有局部扩散的患者也有30%~40%,只有不到20%的患者能进行手术切除[4-5]。因此,寻找早期诊断的生物标志物对于预防和预测PAAD的进展至关重要。除了基因改变外,癌症往往伴随着多种异常的表观遗传修饰改变。DNA甲基化是研究最为深入的表观遗传修饰形式。DNA甲基化异常(高甲基化或低甲基化)主要集中发生在启动子CpG岛区域,可以在不改变基因序列的条件下调控基因的表达[6]。抑癌基因启动子CpG岛异常高甲基化会导致其表达沉默,而癌基因CpG岛的去甲基化会导致其表达上调,从而促进肿瘤进展[7-10]。由于DNA甲基化通常发生在肿瘤早期的体细胞突变之前,且样本容易获取和保存,因此DNA甲基化的鉴定在各种人类癌症的临床前研究中展现出广阔的应用前景。例如,识别患者肿瘤组织或血液中的游离DNA(cell-free DNA,cfDNA)样本的甲基化模式,可以帮助进行肿瘤分型[11-12]。基于关键MDG的表达水平或甲基化水平构建预后模型,可以精准地预测患者的预后[13-15]。总之,甲基化生物标志物检测的广泛应用和表观遗传药物靶点的出现,为癌症的诊断和治疗提供了新的可能性。但到目前为止,在PDAC中尚未发现能够运用于临床的甲基化生物标志物。
中间丝家族孤儿蛋白1(intermediate filament family orphan 1,IFFO1)是中间丝蛋白家族的成员,在大多数真核细胞类型的细胞骨架和核膜中发挥重要作用。既往研究[16]发现,IFFO1主要通过非同源末端连接(non-homologous end joining,NHEJ)途径结合到断裂的DNA末端,从而防止染色体易位和癌症的发生。此外, IFFO1被认为是卵巢癌的新型抑癌分子,能够抑制肿瘤转移和恢复对顺铂的敏感性[17]。另外,IFFO1甲基化水平在卵巢癌患者腹水的cfDNA中异常升高,可作为腹水中肿瘤DNA的标志物[18-19]。然而,目前IFFO1在PAAD中的作用并不清楚。本研究采用PAAD转录组数据与甲基化芯片数据的联合筛选,鉴定出IFFO1可作为PAAD的新型甲基化标志物。随后通过临床标本和体外实验分析IFFO1在PAAD中的表达,以及在PAAD细胞迁移、侵袭中的作用。最后,通过癌症基因组图谱(The Cancer Genome Atlas,TCGA)数据库评估IFFO1甲基化在泛癌中的诊断和预后预测价值。
1 材料和方法
1.1 材料
1.1.1 公共数据来源
从TCGA数据库(https://portal.gdc.cancer. gov/)中获取178例PAAD组织、4例癌旁组织的mRNA表达数据,以及185例PAAD组织、10例癌旁组织的甲基化芯片数据。从高通量基因表达(Gene Expression Omnibus,GEO)数据库(https://www.ncbi.nlm.nih.gov/geo/)中获取16例PAAD组织、8例癌旁组织的甲基化芯片数据(GSE53051),以及183例健康人血液的甲基化芯片数据(GSE69270)。从国际癌症基因组联盟(International Cancer Genome Consortium,ICGC)数据库(https://dcc.icgc.org/)中获取178例PAAD组织及25例癌旁组织的甲基化芯片数据(PACA-AU)。从UCSC Xena数据库(https://xena.ucsc.edu/)中获取18种TCGA泛癌组织及癌旁对照组织的mRNA表达数据及甲基化芯片数据。
1.1.2 组织标本及细胞系来源
27例PAAD患者的癌组织及其对应癌旁组织的病理切片来自上海交通大学医学院附属第一人民医院。本研究涉及的所有样本均通过上海交通大学医学院附属第一人民医院人类伦理委员会的审核批准(伦理编号:院伦审[2017]53号)。PAAD细胞系MIA PaCa2、BxPC-3、AsPC-1、Capan-2和正常胰腺导管上皮细胞系H6C7均购自中国科学院典型培养物保藏委员会细胞库。
1.1.3 主要试剂
IFFO1抗体购自武汉三鹰生物技术有限公司,PV9000免疫组织化学试剂盒购自北京中杉金桥生物技术有限公司,结晶紫、强力抗原修复液均购自上海碧云天生物技术有限公司, DMEM培养基、胎牛血清(fetal bovine serum,FBS)均购自美国Gibco公司,RNA提取试剂盒购自苏州新赛美生物科技有限公司,RNA反转录试剂盒、C115-02同源重组试剂盒及HiScript Ⅲ RT SuperMix均购自南京诺维赞生物科技股份有限公司,实时荧光定量聚合酶链反应(real-time fluorescence quantitative polymerase chain reaction,RTFQ-PCR)试剂盒购自翌圣生物科技(上海)股份有限公司,Opti-MEM减血清培养液、转染试剂LipofectamineTM3000均购自美国Thermo Fisher Scientific公司,4%多聚甲醛溶液、甲醇均购自国药集团化学试剂有限公司,transwell小室购自美国Corning公司,pLenti-CMV-EGFP-3FLAG-PGK-Puro质粒购自和元生物技术(上海)股份有限公司。
1.1.4 主要仪器
生物安全柜、细胞恒温培养箱均购自美国Thermo Fisher Scientific公司,水浴锅购自拓赫机电科技(上海)有限公司,光学显微镜购自日本Nikon公司,摇床振荡器购自普睿博仪器(杭州)有限公司,离心机购自德国Eppendorf公司,QuantStudio5 RTFQ-PCR仪购自美国Applied Biosystems公司。
1.2 方法
1.2.1 甲基化驱动基因(methylation-driven gene,MDG)筛选
具体的筛选路线见图1A。首先分别对4组甲基化芯片数据进行差异甲基化分析得到差异甲基化基因(differentially methylated genes,DMGs),在TCGA mRNA数据中对差异表达基因(differentially expressed genes,DEGs)进行差异表达分析。对于癌组织和癌旁组织的甲基化芯片数据(TCGA-PAAD-meth、PACA-AU、GSE53051),采用R包ChAMP进行数据的读入、探针的过滤和数据标准化和甲基化差异分析,在过滤时仅保留位于启动子区域的CpG探针,差异分析阈值设置为|Δβ-value|>0.2,P.adj<0.05。对于健康人血液的甲基化芯片数据,先将其与TCGA数据库PAAD患者的甲基化芯片数据进行合并,再按上述步骤寻找DMGs。对于癌和癌旁的mRNA数据,将原始mRNA表达矩阵进行log2(FPKM+1)标准化处理,之后采用R包limma进行差异分析,差异分析阈值设置为 |log2 FC|>1,P.adj<0.05。随后,分别将所得高甲基化基因与低表达基因取交集,低甲基化基因与高表达基因取交集从而得到MDG。最后,我们通过生存分析结合单因素COX回归分析进一步筛选潜在的预后相关MDG。
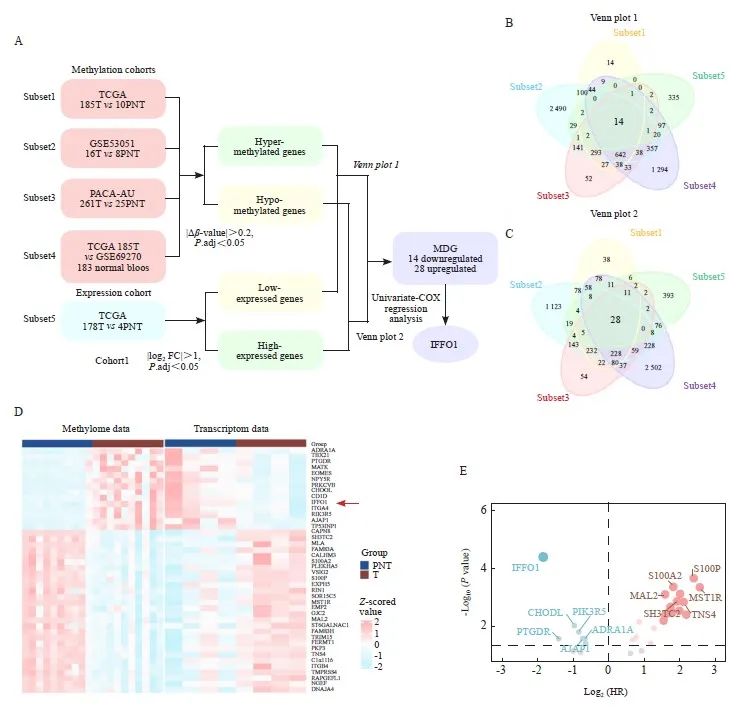
图1 筛选PAAD的MDG
Fig. 1 Screening of MDG in PAAD
A: A flowchart illustrating the data included and the screening strategy employed in this study. B, C: Venn diagrams depicting hypermethylated-downregulated genes (B) and hypomethylated-upregulated genes (C) identified within the gene expression and methylation datasets. D: A heatmap displaying the normalized expression and methylation values of MDG within the TCGA-PAAD cohort. E: Volcano plots demonstrating the prognostic value of MDG.
1.2.2 免疫组织化学
将病理切片置于65 ℃烘箱中烘烤45 min,经二甲苯脱蜡、乙醇梯度水化后,用抗原修复液水浴修复30 min,冷却至室温。采用内源性过氧化物酶阻断剂阻断后加入一抗(IFFO1抗体,工作浓度1∶100)4 ℃温育过夜。加入反应增强液、增强酶标山羊抗小鼠/兔免疫球蛋白G(immunoglobulin G,IgG)聚合物分别温育20 min,采用3,3’-二氨基联苯胺(3,3’-diaminobenzidine,DAB)显色,随后采用苏木精对细胞核进行染色,最后乙醇脱水后滴加中性树脂封固。依据IFFO1染色的深度及面积对结果进行评分,标准如下:① 按染色强度分为0分(无染色)、1分(轻度染色)、2分(中度染色)和3分(强染色);② 按染色面积比例分为0分(0%~5%)、1分(5%~25%)、 2分(25%~50%)、3分(50%~75%)和4分(75%~100%)。最终,一个样本的染色分数为染色面积与染色强度的乘积。
1.2.3 蛋白质印迹法(Western blot)
取适量蛋白样品加入10%的十二烷基硫酸钠聚丙烯酰胺凝胶电泳(sodium dodecylsulphate polyacrylamide gel electrophoresis,SDS-PAGE)胶的泳道内,恒压80 V进行电泳,后于 400 mA/90 min条件下转膜,采用5%脱脂奶粉室温封闭3 h,加入适量一抗(IFFO1抗体, 1∶1 000)4 ℃冰箱内温育过夜;用含有吐温-20三乙醇胺缓冲盐溶液(tris-buffered saline Tween,TBST)洗涤后加入二抗(1∶5 000)室温温育1 h,再次用TBST洗涤。按1∶1比例配制发光显影液,使用化学发光检测仪进行曝光。
1.2.4 RTFQ-PCR
收集所需检测的细胞,采用RNA抽提试剂盒提取总RNA,随后使用HiScript Ⅲ RT SuperMix将所得RNA反转录为cDNA,并用焦碳酸二乙酯(diethyl pyrocarbonate,DEPC)水将产物稀释5倍。随后,在96孔板中配置RTFQ-PCR体系:每孔20 μL,包含SYBR Green Master Mix 10 μL,上下游引物(10 μmol/L)各0.4 μL,模版cDNA 2 μL,DEPC水7.2 μL。随后在QuantStudio5仪器上进行PCR实时扩增。采用2-ΔΔCt法计算基因的相对表达量。IFFO1的正向引物序列为5’-TCCACCACCTATTCCTCGTCG-3’,反向引物序列为5’-CTCCGCTTGTACTCGTCCC-3’;GAPDH的正向引物序列为5’-GGAGCGAGA TCCCTCCAAAAT-3’,反向引物序列为5’-GGC TGTTGTCATACTTCTCATGG-3’。
1.2.5 过表达引物设计及质粒构建
IFFO1过表达的正向引物序列为5’-TCGAGC TCAAGCTTCGAATTCATGAATCCGTTATTCGG CCC-3’,反向引物序列为5’-GTCATCCTTGTAG TCGGATCCCTATCTCATGGAGCTGTCAGATG AG-3’。
按照同源重组试剂盒说明进行质粒构建:采用PCR在模版cDNA中扩增IFFO1 CDS序列。对质粒载体进行4 ℃水浴酶切30 min,酶切位点为EcorI和BamHI。将所得扩增产物与酶切产物进行琼脂糖电泳,条件为80 V/60 min。对跑胶产物进行切胶回收纯化。将纯化后的克隆载体与插入片段按照说明书推荐比例进行单片段同源重组反应。取1 μL重组产物与50 μL FastT1感受态细胞混合后置于冰上30 min,随后42 ℃水浴30 s,均匀涂布于LB平板上。第2天挑取克隆送测序,结果返回后采用SnapGene软件进行序列比对。
1.2.6 转染
提前1 d铺板,待细胞融合度为70%~90%时,更换为无血清培养基。使用Opti-MEM分别稀释相关质粒DNA和LipofectamineTM3000。将质粒DNA混合液缓慢滴入LipofectamineTM3000混合液中,静置15 min。将上述液体混合后加入6孔板中,前后轻摇细胞板混合均匀。转染6~8 h后更换新鲜培养基,并展开后续相关功能实验。
1.2.7 细胞划痕实验
将转染后24 h的细胞重新消化均匀铺于6孔板中使其贴壁完全后密度接近100%,使用100 μL移液器吸嘴刮划贴壁细胞,采用磷酸缓冲盐溶液(phosphate-buffered saline,PBS)洗涤1遍后,更换为无血清培养基继续培养24 h,然后在光学显微镜下进行拍照,并计算最后愈合率。
1.2.8 细胞迁移实验
将transwell置于24孔板中,将转染后24 h的细胞重新消化离心,使用无血清DMEM重悬并计数1×105个细胞铺于小室的上室,下室加入500 μL含有10% FBS的DMEM培养基。培养48 h后,采用4%多聚甲醛溶液室温固定细胞 20 min,结晶紫染色30 min,使用棉签擦去小室上室的细胞,于显微镜下随机选择4个视野,拍照并计数。
1.3 统计学处理
使用R语言(4.3.1版本)及GraphPad Prism 9.0软对数据进行统计分析及可视化。非配对数据两两比较采用独立样本t检验,配对数据两两比较采用配对样本t检验,两组以上比较采用Kruskal-Wallis检验。采用对数秩检验进行Kaplan Meier生存分析,采用COX比例风险回归模型进行单因素预后分析。Pearson相关性分析用来计算基因表达与甲基化水平的相关性。采用R包clusterProfier中的两种不同富集分析方法[超几何分布检验[20]和基因集富集分析(gene set enrichment analysis,GSEA)算法[21]]进行富集分析。使用R包pROC进行受试者工作特征(receiver operating characteristic,ROC)曲线的绘制和曲线下面积(area under curve,AUC)的计算。AUC越接近1说明预测诊断效能越好。P<0.05为差异有统计学意义。
2 结 果
2.1 PAAD的MDG筛选结果
为了筛选PAAD的MDG,从TCGA数据库、 ICGC数据库及GEO数据库中获取PAAD组织及癌旁组织的mRNA表达数据(TCGA-PAAD-mRNA)、DNA甲基化芯片数据(TCGA-PAAD-meth、GSE53051、PACA-AU)以及健康人血液的DNA甲基化芯片数据(GSE69270)。随后,分别在每组数据中进行差异甲基化分析及差异表达分析(图1A)。通过对上述数据的交叉筛选,最终鉴定得到了在PAAD中显著高甲基化、在正常人血液中及癌旁组织中低甲基化同时表达下调的基因14个,以及在PAAD中显著低甲基化、在正常人血液中及癌旁组织中高甲基化同时表达上调的基因28个,交叉筛选结果见图1B、1C。TCGA数据库中配对PAAD与癌旁患者候选MDG的甲基化与表达水平见图1D。为进一步明确候选MDG与PAAD患者预后的关系,我们进行了Kaplan-Meier生存分析及单因素COX回归分析,并依据结果的风险比(hazard ratio,HR)和P值绘制了火山图,结果见图1E,多数低甲基化而异常高表达的基因与PAAD患者的不良预后相关,高甲基化而低表达的基因与PAAD患者的良好预后相关。其中,IFFO1的P值最显著,HR值最小(HR=0.28,P<0.001),据此可以推测其与PAAD患者的预后关系较为密切。鉴于IFFO1在PAAD中的预后价值及作用尚未见报道,因此本研究选择IFFO1作为后续验证的基因。
2.2 IFFO1高甲基化低表达与PAAD患者不良预后相关
不同数据集中PAAD组织及癌旁组织的IFFO1表达水平及启动子甲基化水平见图2A。为进一步明确IFFO1基因的表达水平与其甲基化水平的关系,我们分析了TCGA数据库中的IFFO1表达水平与IFFO1甲基化水平的相关性,结果见图2B,IFFO1的表达与IFFO1的启动子平均甲基化水平以及IFFO1启动子区域的多个CpG探针的甲基化水平均具有显著的负相关关系(r<-0.5,P<0.05)。我们进一步利用免疫组织化学法在27例PAAD患者的癌组织及相应癌旁组织的病理切片中验证了IFFO1的表达,结果见图2C,IFFO1在PAAD的癌旁组织中的免疫组织化学评分显著高于癌组织(P<0.05)。为明确IFFO1的表达与临床病理学特征的关系,我们比较了TCGA数据库中不同分级、分期PAAD患者的IFFO1表达水平。结果显示,IFFO1的表达量随肿瘤分级和分期的增加而降低(图2D)。同时,Kaplan-Meier生存分析提示,IFFO1低表达或高甲基化患者,具有更短的总生存期(图2E)。综合以上结果推断,在PAAD中IFFO1发生启动子异常高甲基化而低表达,与PAAD的不良预后相关。
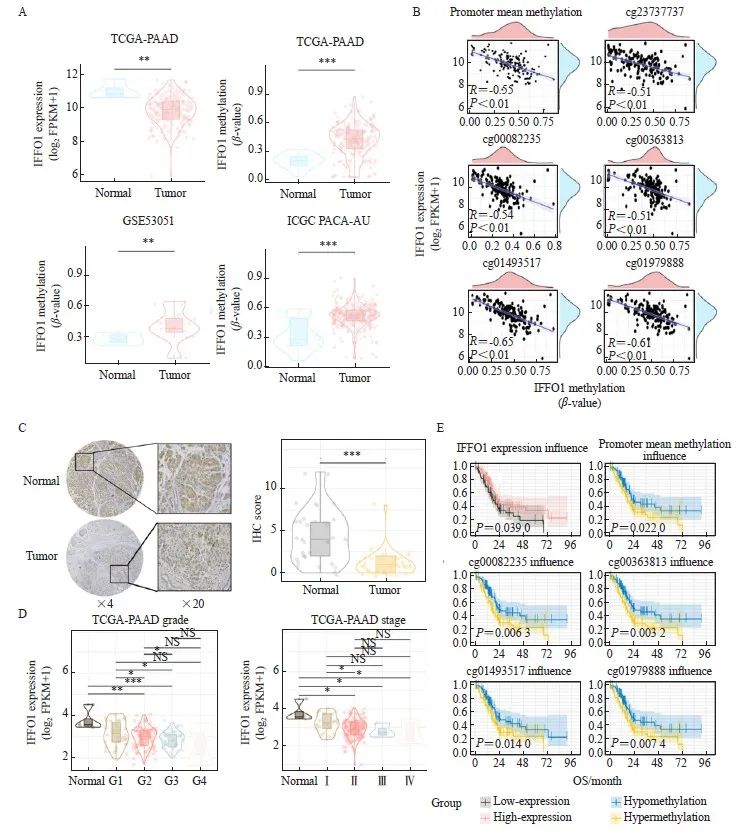
图2 IFFO1高甲基化低表达与PAAD患者不良预后相关
Fig. 2 Hypermethylation and low IFFO1 expression correlate with poor prognosis in PAAD patients
A: Comparison of methylation levels and expression levels of IFFO1 in adjacent normal tissues versus tumor tissues from the TCGA database. B: The correlation between IFFO1 expression levels and IFFO1 promoter methylation. C: Immunohistochemical analysis of IFFO1 expression in adjacent normal versus tumor biopsies of PAAD. D: Distribution of IFFO1 expression across different tumor grades (E) and stages (F) as reported in the TCGA database. E: Kaplan-Meier survival analysis of IFFO1 expression from the TCGA database, illustrating differences in survival outcomes based on varying IFFO1 expression or methylation levels. OS: Overall survival; NS: No significance. *: P<0.05; **: P<0.01; ***: P<0.001.
2.3 富集分析
为进一步探究IFFO1在PAAD发生、发展中所参与的生物学过程及作用,我们在TCGA数据库中,依据IFFO1的中位表达水平将PAAD分为高表达组和低表达组并进行了差异分析,与IFFO1最相关的正负各20个差异基因见图3。我们随后将与IFFO1正相关同时差异倍数最大的前200个基因进行了基因本体论(gene ontology,GO):生物学过程(biological process,BP)富集分析,结果见图3B,这些基因主要包含一些细胞骨架蛋白,参与细胞外基质的重塑,同时参与细胞的迁移和运动的负向调控。随后,我们对全部基因按照log2 FC从大到小排序进行GSEA富集分析(图3C),结果同样提示迁移和运动负向调控通路主要在IFFO1高表达患者中富集(NES>2,P<0.001)。因此,我们推断IFFO1可能抑制胰腺肿瘤细胞的侵袭转移过程。
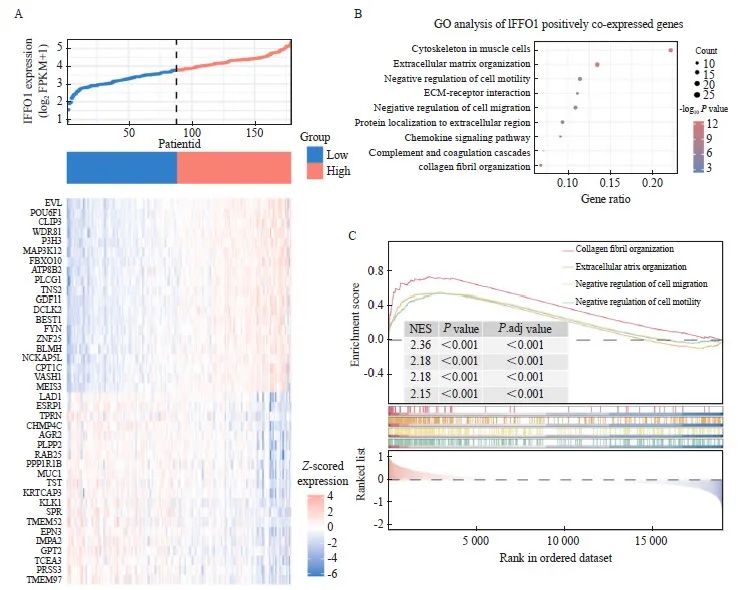
图3 TCGA数据库中IFFO1相关GSEA和GO富集分析
Fig. 3 GSEA and GO enrichment analysis of IFFO1 in the TCGA database
A: The top 20 upregulated and downregulated co-expressed genes associated with IFFO1 expression in PAAD. B: GO enrichment analysis highlighting the biological processes significantly linked to IFFO1. C: GSEA illustrating the pathways associated with IFFO1 expression.
2.4 IFFO1过表达抑制PAAD细胞株转移
为进一步明确IFFO1的表达对PAAD细胞株迁移和侵袭的影响。我们首先利用Western blot和RTFQ-PCR技术检测了不同PAAD细胞系 AsPC-1、BXPC-3、Capan-2、MIA PaCa2,以及正常胰腺细胞系H6C7中IFFO1的表达情况,结果见图4A、4B,在PAAD细胞系中IFFO1的蛋白质和mRNA表达水平均较正常胰腺导管上皮细胞株H6C7显著降低。随后,我们构建了IFFO1过表达质粒,并向ASPC-1和Capan-2细胞系中分别转染了IFFO1过表达质粒以及对照空载质粒。我们利用Western blot验证IFFO1的过表达效率,结果显示,与对照组相比,转染过表达质粒后, IFFO1表达显著增加(图4C),提示转染成功。随后,我们分别采用细胞划痕实验、细胞侵袭实验检测上述已转染的AsPC-1和Capan-2细胞株的迁移和侵袭能力,结果显示,在两种细胞株中,转染IFFO1-OE质粒的PAAD细胞的迁移和侵袭能力均显著低于转染空载质粒的PAAD细胞(P均<0.01,图4D、4E)。上述结果表明IFFO1过表达可抑制PAAD细胞的侵袭和迁移能力。
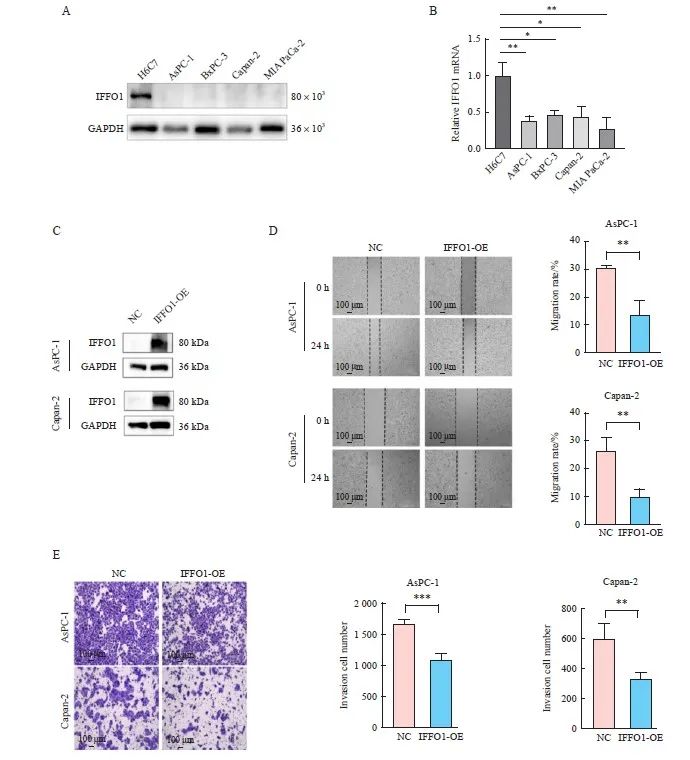
图4 过表达IFFO1抑制AsPC-1和Capan-2细胞的迁移和侵袭能力
Fig. 4 Overexpression of IFFO1 inhibits the migration and invasion ability of AsPC-1 and Capan-2 cells
A, B: Expression levels of IFFO1 in various PAAD cell lines were assessed using Western blot (A) and RTFQ-PCR (B). C: The IFFO1 expression levels in AsPC-1 and Capan-2 cells, transfected with either a control vector or IFFO1 overexpression (IFFO1-OE) vector, were measured by Western blot. D-E: The impact of IFFO1 overexpression on the migration (D) and invasion (E) capabilities of AsPC-1 and Capan-2 cells was evaluated. *: P<0.05; **: P<0.01; ***: P<0.001.
2.5 IFFO1甲基化可作为多种实体肿瘤患者预后不良的标志物
为进一步明确IFFO1甲基化的诊断和预后效能,我们收集了来自TCGA泛癌队列18种不同癌组织及相应癌旁组织的甲基化数据及表达数据,包括405例膀胱尿路上皮癌(bladder urothelial carcinoma,BLCA)、1 072例乳腺癌(breast invasive carcinoma,BRCA)、296例宫颈鳞癌和腺癌(cervical squamous cell carcinoma and endocervical adenocarcinoma,CESC)、36例胆管癌(cholangiocarcinoma,CHOL)、453例结肠癌(colon adenocarcinoma,COAD)、152例食管癌(esophageal carcinoma,ESCA)、144例星形胶质瘤(glioblastoma multiforme,GBM)、495例头颈鳞状细胞癌(head and neck squamous cell carcinoma,HNSC)、526例肾透明细胞癌(kidney renal clear cell carcinoma,KIRC)、369例肝细胞肝癌(liver hepatocellular carcinoma, LIHC)、510例肺腺癌(lung adenocarcinoma,LUAD)、496例肺鳞癌(lung squamous cell carcinoma,LUSC)、178例PAAD、481例前列腺癌(prostate adenocarcinoma,PRAD)、163例直肠腺癌(rectum adenocarcinoma,READ)、 373例胃癌(stomach adenocarcinoma,STAD)、497例甲状腺癌(thyroid carcinoma,THCA)和537例子宫内膜癌(uterine corpus endometrial carcinoma,UCEC)组织样本。我们评估了IFFO1在18种不同的癌组织及相应癌旁组织中的表达水平和甲基化水平差异,结果显示, IFFO1在多个癌种(如COAD、PAAD、CHOL、UCEC、BLCA、PRAD、LUAD、BRCA、LUSC、THCA)中显著高甲基化而低表达(图5A、5B)。同时,相关性分析结果所示,在多数癌症中,IFFO1启动子CpG探针的甲基化水平与IFFO1的表达水平呈显著负相关关系 (P<0.05,图5C)。为明确IFFO1甲基化的诊断价值,我们绘制了ROC曲线,根据ROC曲线的AUC,IFFO1甲基化水平对于COAD ( AUC=0.778)、PAAD( AUC=0.868)、CHOL(AUC=0.890)、UCEC(AUC=0.983)、PRAD(AUC=0.918)、LUAD(AUC=0.962)、BRCA(AUC=0.901)及LUSC(AUC=0.972)的诊断均具有较高的灵敏度和特异度(图5D)。同时,为明确IFFO1甲基化的预后预测价值,我们利用Kaplan-Meier生存分析检测了IFFO1甲基化水平对各类癌症患者总生存期的影响,结果显示,除PAAD外,IFFO1高甲基化还与LUSC、UCEC、COAD及BLCA的不良预后有关(图5E)。上述结果说明在多种癌症中,IFFO1甲基化可以作为潜在的诊断和预后标志物。
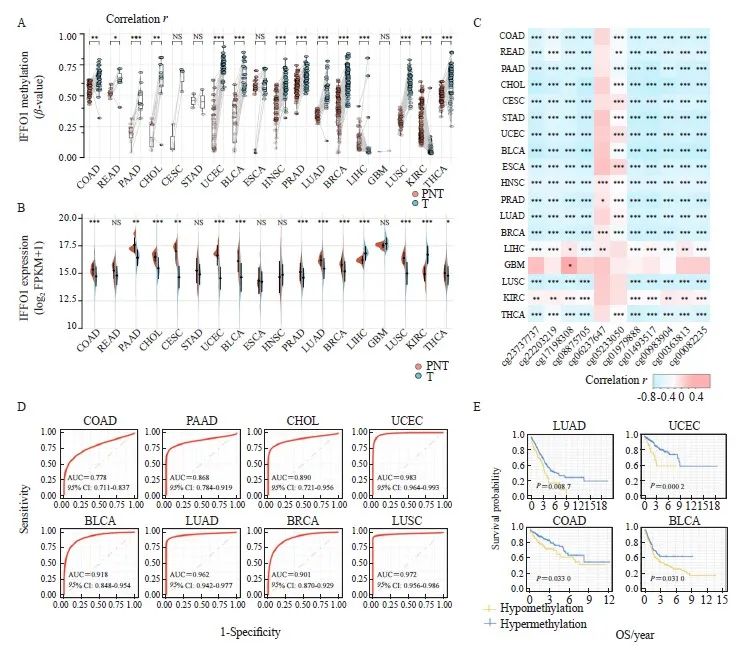
图5 IFFO1异常甲基化在泛癌的诊断和预后预测中的价值
Fig. 5 Diagnostic and prognostic prediction value of IFFO1 aberrant methylation in Pan-Cancer
A, B: Comparative analysis of IFFO1 methylation levels (A) and gene expression levels (B) in adjacent normal tissues versus tumor tissues across various cancer types, as obtained from the TCGA database. C: A heatmap illustrating the correlation between IFFO1 gene expression and the methylation status of promoter CpG sites. D: ROC curve analysis assessing the diagnostic potential of IFFO1 promoter methylation across different tumor types. E: Kaplan-Meier plots demonstrating the prognostic significance of IFFO1 promoter methylation in various tumor types found in the TCGA database. *: P<0.05; **: P<0.01; ***: P<0.001.
3 讨 论
PAAD是一种起病隐匿、早期诊断困难的消化道恶性肿瘤。目前在临床上主要结合病理学分级及血清标志物[如糖类抗原19-9(carbohydrate antigen 19-9,CA19-9)、糖类抗原12-5(carbohydrate antigen 12-5,CA12-5)、癌胚抗原(carcinoembryonic antigen,CEA)]对疾病进行大致评估,其准确度仍有待提高[22-23]。因此,发现新的PAAD诊断标志物至关重要。近年来,癌症中的DNA甲基化改变被视为强有力的诊断及预后生物标志物,因为其具有在癌变早期出现、随着时间的推移在固定样品和体液中相对稳定、易于检测等优势[24-26]。然而,在PAAD中尚缺乏甲基化相关诊断的预后标志物。
通过甲基化芯片数据和mRNA表达谱数据的联合挖掘,我们成功地鉴定出了PAAD的新型MDG IFFO1。IFFO1是中间丝家族蛋白的一员,该家族蛋白是真核生物细胞骨架蛋白的重要成员之一。IFFO1最先在一项质谱的相互作用分析研究[27]中被鉴定,该研究发现IFFO1可以与核心DNA修复因子CXC族趋化因子受体4(CXC chemokine receptor 4,CXCR4)相互作用,在DNA双链断裂的NHEJ修复中发挥重要作用,后续研究进一步证实,在野生型细胞中,IFFO1可以充当支架,允许XRCC4和核纤层蛋白lamin A/C组装成复合物,从而确保断裂的DNA末端保持在原始位置,以促进基因组完整性的维持。当IFFO1表达缺失,导致断裂的DNA末端可以在细胞核周围自由移动,从而可能导致致癌染色体易位和肿瘤的发生[16,28]。此外,另有实验研究证明,在卵巢癌中IFFO1受到转录水平m6A RNA甲基化及转录后水平HDAC5/YY1去乙酰化的双重调控而表达下调。IFFO1的过表达可以抑制β-catenin的核转位,减少肿瘤转移和耐药性[17]。上述研究结果表明,IFFO1可以通过多种机制抑制癌症的发生、发展。本研究发现,在PAAD中IFFO1受到DNA启动子甲基化调控,且表达水平与多个启动子CpG位点甲基化水平呈现显著负相关关系。为进一步明确IFFO1在PAAD中的表达以及与患者预后的关系,我们对27例PAAD及癌旁组织的病理切片进行了IHC染色分析,发现IFFO1在PAAD组织中低表达。同时, TCGA数据库分析提示,IFFO1表达随患者的分级和分期的增加而降低,高甲基或低表达IFFO1的患者总生存率更差。随后,我们利用公共数据库进行了富集分析,GSEA和GO富集分析结果均提示在PAAD中IFFO1可以抑制PAAD细胞的运动和迁移。我们在PAAD细胞系AsPC-1和Capan-2中过表达IFFO1后发现细胞的迁移和侵袭能力显著下调。最后,为明确IFFO1甲基化作为PAAD诊断指标的特异性,我们进行了泛癌甲基化分析,结果提示,IFFO1甲基化在多种癌症中均具有良好的诊断能力并与患者预后相关。
综上所述,本研究探讨了在PAAD中,IFFO1基因甲基化及表达水平与患者预后的关系以及IFFO1与PAAD细胞侵袭、转移表型的关系。IFFO1甲基化可作为PAAD患者诊断和预后预测的新型标志物。
利益冲突声明:所有作者均声明不存在利益冲突。
作者贡献声明:
徐梓淇:实验设计和验证,生物信息学分析,撰写和修改文章;
胡睿智:实验设计和验证;
李军建:实验设计,实验操作指导;
王红霞:实验设计,撰写和修改文章;
桑友洲:实验设计,撰写和修改文章。
[参考文献]
[1] SIEGEL R L, MILLER K D, WAGLE N S, et al. Cancer statistics, 2023[J]. CA Cancer J Clin, 2023, 73(1): 17-48.
[2] HE S Y, XIA C F, LI H, et al. Cancer profiles in China and comparisons with the USA: a comprehensive analysis in the incidence, mortality, survival, staging, and attribution to risk factors[J]. Sci China Life Sci, 2024, 67(1): 122-131.
[3] WANG Y A, YAN Q J, FAN C M, et al. Overview and countermeasures of cancer burden in China[J]. Sci China Life Sci, 2023, 66(11): 2515-2526.
[4] 李天骄, 叶龙云, 金凯舟, 等. 2023年度胰腺癌研究及诊疗新进展[J].中国癌症杂志, 2024, 34(1): 1-12.
LI T J, YE L Y, JIN K Z, et al. Advances in basic research, clinical diagnosis and treatment of pancreatic cancer in 2023[J]. China Oncol, 2024, 34(1): 1-12.
[5]HALBROOK C J, LYSSIOTIS C A, PASCA DI MAGLIANO M, et al. Pancreatic cancer: advances and challenges[J]. Cell, 2023, 186(8): 1729-1754.
[6]PORTELA A, ESTELLER M. Epigenetic modifications and human disease[J]. Nat Biotechnol, 2010, 28(10): 1057-1068.
[7]EHRLICH M. DNA hypomethylation in cancer cells[J]. Epigenomics, 2009, 1(2): 239-259.
[8]ZHAO Y H, YANG M, WANG S J, et al. An overview of epigenetic methylation in pancreatic cancer progression[J]. Front Oncol, 2022, 12: 854773.
[9]GOTOH M, ARAI E, WAKAI-USHIJIMA S, et al. Diagnosis and prognostication of ductal adenocarcinomas of the pancreas based on genome-wide DNA methylation profiling by bacterial artificial chromosome array-based methylated CpG island amplification[J]. J Biomed Biotechnol, 2011, 2011: 780836.
[10]BAYLIN S B, OHM J E. Epigenetic gene silencing in cancer–a mechanism for early oncogenic pathway addiction?[J]. Nat Rev Cancer, 2006, 6: 107-116.
[11]HU J, OTHMANE B, YU A Z, et al. 5mC regulator-mediated molecular subtypes depict the hallmarks of the tumor microenvironment and guide precision medicine in bladder cancer[J]. BMC Med, 2021, 19(1): 289.
[12]CHEMI F, PEARCE S P, CLIPSON A, et al. cfDNA methylome profiling for detection and subtyping of small cell lung cancers[J]. Nat Cancer, 2022, 3(10): 1260-1270.
[13]XIAO M J, LIANG X J, YAN Z M, et al. A DNA-methylation-driven genes based prognostic signature reveals immune microenvironment in pancreatic cancer[J]. Front Immunol, 2022, 13: 803962.
[14]PENG Y J, WU Q Y, WANG L X, et al. A DNA methylation signature to improve survival prediction of gastric cancer[J]. Clin Epigenetics, 2020, 12(1): 15.
[15]ZHANG M, WANG Y L, WANG Y, et al. Integrative analysis of DNA methylation and gene expression to determine specific diagnostic biomarkers and prognostic biomarkers of breast cancer[J]. Front Cell Dev Biol, 2020, 8: 529386.
[16]LI W, BAI X Z, LI J, et al. The nucleoskeleton protein IFFO1 immobilizes broken DNA and suppresses chromosome translocation during tumorigenesis[J]. Nat Cell Biol, 2019, 21(10): 1273-1285.
[17]ZHANG Y, QIU J G, JIA X Y, et al. METTL3-mediated N6-methyladenosine modification and HDAC5/YY1 promote IFFO1 downregulation in tumor development and chemo-resistance[J]. Cancer Lett, 2023, 553: 215971.
[18]WERNER B, SJOQUIST K M, ESPINOZA D, et al. Cell-free DNA in plasma and ascites as a biomarker of bevacizumab response- a translational research sub-study of the REZOLVE (ANZGOG-1101) clinical trial[J]. Transl Oncol, 2024, 43: 101914.
[19]WERNER B, YUWONO N, DUGGAN J, et al. Cell-free DNA is abundant in ascites and represents a liquid biopsy of ovarian cancer[J]. Gynecol Oncol, 2021, 162(3): 720-727.
[20]YU G C, WANG L G, HAN Y Y, et al. clusterProfiler: an R package for comparing biological themes among gene clusters[J]. OMICS, 2012, 16(5): 284-287.
[21]SUBRAMANIAN A, TAMAYO P, MOOTHA V K, et al. Gene set enrichment analysis: a knowledge-based approach for interpreting genome-wide expression profiles[J]. Proc Natl Acad Sci USA, 2005, 102(43): 15545-15550.
[22]HUMPHRIS J L, CHANG D K, JOHNS A L, et al. The prognostic and predictive value of serum CA19.9 in pancreatic cancer[J]. Ann Oncol, 2012, 23(7): 1713-1722.
[23]ASAOKA T, MIYAMOTO A, MAEDA S, et al. Prognostic impact of preoperative NLR and CA19-9 in pancreatic cancer[J]. Pancreatology, 2016, 16(3): 434-440.
[24]KOCH A, JOOSTEN S C, FENG Z, et al. Analysis of DNA methylation in cancer: location revisited[J]. Nat Rev Clin Oncol, 2018, 15(7): 459-466.
[25]LAIRD P W. The power and the promise of DNA methylation markers[J]. Nat Rev Cancer, 2003, 3(4): 253-266.
[26]BAYLIN S B, JONES P A. A decade of exploring the cancer epigenome-biological and translational implications[J]. Nat Rev Cancer, 2011, 11(10): 726-734.
[27]CABELLO-LOBATO M J, JENNER M, CISNEROS-AGUIRRE M, et al. Microarray screening reveals two non-conventional SUMO-binding modules linked to DNA repair by non-homologous end-joining[J]. Nucleic Acids Res, 2022, 50(8): 4732-4754.
[28]GHOSH D, RAGHAVAN S C. Nonhomologous end joining: new accessory factors fine tune the machinery[J]. Trends Genet, 2021, 37(6): 582-599.