引言
肿瘤微生物组(tumor microbiome)对肿瘤生物学和治疗响应的影响已逐渐成为科研的热点。近年来,研究表明肿瘤微生物群可能与肿瘤发生、发展及其微环境中的免疫调节有密切关联。特别是,肿瘤微生物组的存在不仅影响肿瘤的生物行为,还可能影响患者对某些化疗药物的反应,如已有研究指出,某些肿瘤内的微生物能够调节肿瘤对吉西他滨(gemcitabine)的抗药性。4月9日发表于Cell杂志的研究“A pan-cancer analysis of the microbiome in metastatic cancer”专注于转移性肿瘤(metastatic cancers)中的肿瘤微生物组,探索其在癌症生物学中可能的作用以及作为潜在治疗靶标的可能性。
该研究采用了高通量测序技术(high-throughput sequencing)来分析肿瘤组织中的微生物群组成,样本来源于全球不同地区的转移性肿瘤样本,这为研究人员提供了独特的视角来观察和分析肿瘤微生物组的多样性和功能。
研究人员在研究中提供了大量的数据和资源,这些资源将有助于未来进行假设驱动的实验(hypothesis-driven experiments),从而更好地理解肿瘤微生物组的复杂作用。尽管该研究结果目前仅为相关性的(correlative),但该数据为将来解析微生物组与肿瘤或免疫细胞共培养(co-cultures)中因果关系提供了基础。

Highlights
在广泛的癌症转移样本中,发现了肿瘤内细菌的DNA,这表明微生物组的存在是普遍的现象。研究还展示了通过组装肿瘤来源的细菌DNA,可以实现对细菌物种的基因组水平的鉴定。
该研究揭示了微生物多样性与肿瘤浸润性中性粒细胞(tumor-infiltrating neutrophils)之间的关联,并发现与细胞和分子肿瘤免疫特性(cellular and molecular tumor immunity features)相关。
在非小细胞肺癌(NSCLC)的患者中,梭状芽孢杆菌(Fusobacterium)的DNA丰度与免疫疗法(immunotherapy)响应不良相关联,提示这种细菌可能是影响治疗效果的一个生物标志物(biomarker)。
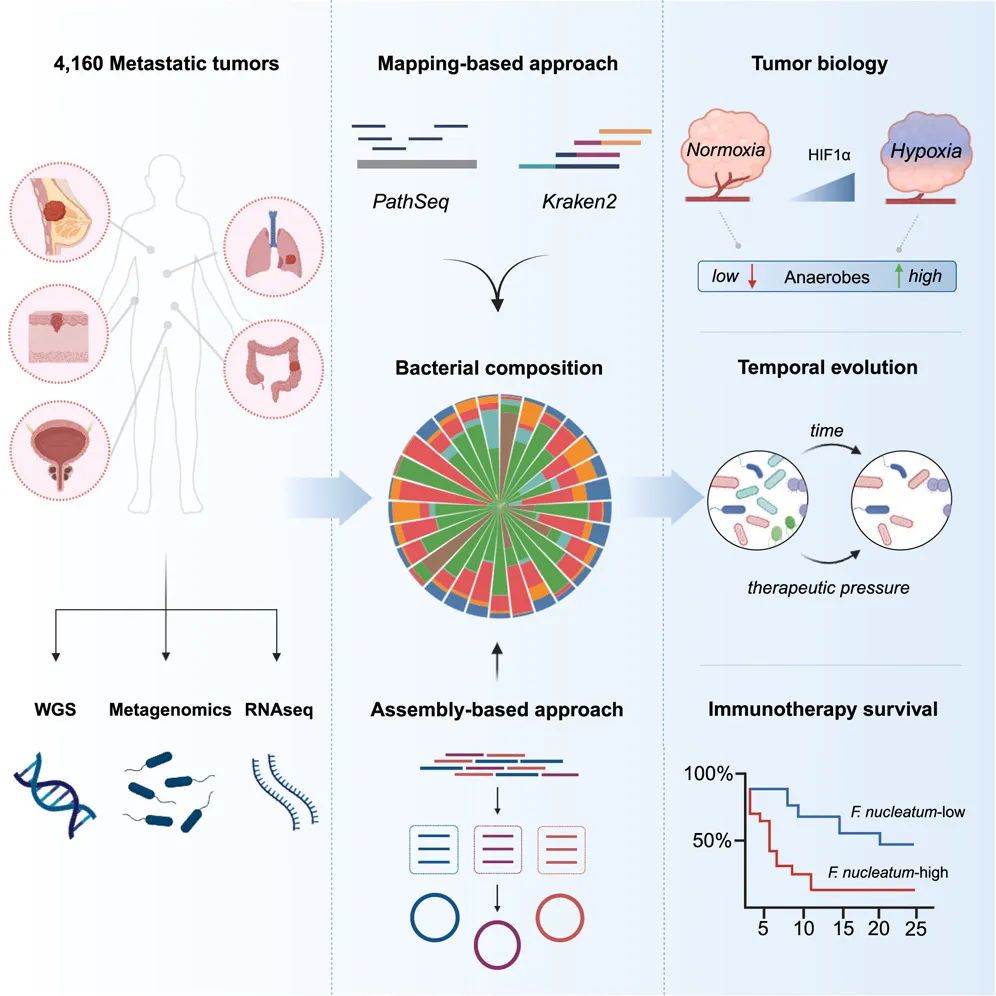
研究流程图(Credit: Cell)
Strategies
该研究通过整合基因组学、转录组学和宏基因组学方法来分析和揭示转移性癌症中微生物组的特征及其对肿瘤微环境(Tumor Microenvironment, TME)的影响。研究中使用了一系列的生物信息学工具来处理和分析数据,旨在深入了解微生物如何在癌症转移过程中发挥作用,以及它们如何可能影响肿瘤对免疫检查点阻断(Immune Checkpoint Blockade, ICB)等治疗的响应。
样本收集与数据整合:研究分析了4160个转移性肿瘤活检样本,包括多种癌症类型和来源于26种组织的数据。通过将宏基因组学、基因组学、转录组学和临床数据进行整合,以形成一个全面的数据集,用于后续分析。
宏基因组学分析(Metagenomics):使用Kraken2和PathSeq这两种计算方法从肿瘤样本中提取未映射读段(unmapped reads),并进行物种水平的基因组组装,以识别和分类肿瘤中的微生物DNA。这一步骤帮助研究者确定肿瘤中存在的微生物种类及其丰度。
基因组和转录组剖析:研究进一步利用基因组数据来分析癌症样本的遗传变异,以及转录组数据来评估与癌症及微生物相互作用相关的基因表达模式。这包括使用基因集富集分析(Gene Set Enrichment Analysis, GSEA)来识别与低氧(Hypoxia)等微环境因素相关的基因表达变化。
数据分析和统计处理:通过比较微生物多样性与肿瘤免疫渗透特性的关系,研究揭示了肿瘤驻留细菌与宿主免疫系统之间的复杂交互作用。研究还探讨了微生物多样性如何与肿瘤对免疫治疗的反应性相关联。
长期跟踪和治疗效果评估:研究还包括对接受不同治疗策略(如免疫治疗、靶向治疗)患者肿瘤样本的纵向采样,分析时间跨度内微生物群落的动态变化及其对治疗的影响。
Behind the Scenes
微生物群落与肿瘤标志物的关联
低氧环境(Hypoxia)与微生物群落
研究表明,肿瘤的低氧环境与厌氧菌(anaerobic bacteria)的丰富度有显著相关性。在肿瘤微环境(Tumor Microenvironment, TME)中,低氧条件不仅是肿瘤生长的一个重要特征,也为厌氧菌提供了适宜的生存环境。通过使用标志性低氧基因(hallmark hypoxia gene signature)对肿瘤样本进行测试,研究人员发现,具有较高厌氧菌多样性的肿瘤在低氧基因集的表达上也更为活跃。这种关联暗示了厌氧菌可能在调节或反映低氧状态中发挥作用。
炎症(Inflammation)与微生物群落
研究还发现,肿瘤中的微生物多样性与炎症相关途径的活化密切相关。通过基因集富集分析(Gene Set Enrichment Analysis, GSEA),研究指出肿瘤中细菌多样性高的样本中,细胞外基质(Extracellular Matrix, ECM)组织和抗微生物肽(Antimicrobial Peptides, AMPs)的相关途径得到了强化。这表明,微生物群落通过影响炎症反应途径,可能参与调控肿瘤生物学行为和肿瘤微环境的动态平衡。
免疫细胞浸润与微生物群落
肿瘤内的微生物群落不仅与炎症和低氧状态有关,还与肿瘤内免疫细胞的浸润程度相关联。研究发现,微生物多样性与癌相关成纤维细胞(Cancer-Associated Fibroblasts, CAFs)的浸润以及免疫排除(immune exclusion)有正相关。此外,高微生物多样性的肿瘤显示出由中性粒细胞、自然杀伤细胞(Natural Killer, NK cells)、巨噬细胞及调节性T细胞(Regulatory T cells, Tregs)驱动的免疫细胞富集,这强调了微生物群落在肿瘤免疫调节中的潜在作用。
免疫治疗效果与微生物群落
微生物群落如何影响免疫治疗的效果,特别是在接受免疫检查点阻断(Immune Checkpoint Blockade, ICB)治疗的非小细胞肺癌(Non-Small Cell Lung Cancer, NSCLC)患者中,富含梭杆菌(Fusobacterium)的微生物组预示着较差的治疗反应。通过比较治疗前后的细菌丰度变化,研究揭示了ICB治疗能显著减少细菌多样性,这一现象在反应良好的患者中尤为明显,暗示微生物组的组成可能预测免疫治疗的效果。
微生物群落与解剖位置的关系及其对肿瘤转移的影响
解剖位置的微生物群落组成
研究表明,不同解剖位置的肿瘤内部存在微生物群落组成的差异。例如,在结直肠癌转移到肝脏的样本中,观察到微生物群落与原发肿瘤部位相比有明显不同的组成。这种差异可能反映了微生物对其所在解剖位置的器官特异性微环境的适应性。通过对多个癌症类型和转移部位的比较,研究发现了特定的微生物群落组合,类似于肠道中的“肠型”(enterotypes)。
微生物对器官特异性微环境的适应性
微生物群落的适应性表现在它们能够在特定的解剖环境中生存并展示出特有的生物学特征。例如,肿瘤转移到肝脏的情况下,肝脏的高血流量和丰富的营养物质为某些微生物提供了繁殖的优势环境。此外,微生物可能通过改变肿瘤微环境中的pH值或氧气浓度,以适应并影响其生存的局部环境。
微生物群落对癌症类型和转移部位的影响
微生物的存在不仅可以影响肿瘤的生长和进展,还可能与肿瘤的转移模式相关。例如,某些细菌如梭杆菌(Fusobacterium)在结直肠癌中的丰富度与肿瘤转移到肝脏的倾向有关。研究还显示,微生物群落的组成可以影响肿瘤细胞的侵袭性和迁移能力,从而可能影响癌症的转移路径。通过分析微生物群落与肿瘤细胞间的相互作用,可以进一步揭示它们如何共同作用于癌症的转移和发展。
微生物群落随时间和治疗介入的变化分析
免疫治疗(Immunotherapy)前后的微生物多样性变化
研究表明,在接受免疫治疗前后,肿瘤微生物群落的多样性会发生显著变化。具体来说,研究对33例接受免疫治疗的患者进行了两次活检,一次在治疗前,一次在治疗后。结果显示,在接受免疫治疗的患者中,治疗后的肿瘤微生物多样性显著下降。这一发现说明,免疫治疗可能通过影响微生物群落的结构来发挥作用,进而改变肿瘤的生物行为。
免疫治疗对特定微生物的影响
研究进一步分析了免疫治疗对特定微生物种类的影响。例如,研究发现,对于那些免疫治疗有反应的患者,肿瘤中的Actinomyces、Bacteroidetes 和 Prevotella 等细菌明显减少。这表明这些微生物可能在调节免疫反应中扮演了一定的角色,可能是通过它们的肽段被肿瘤细胞的HLA(人类白细胞抗原)机制呈递,引起细胞毒性免疫反应。
与治疗反应相关的微生物指标
此外,研究还发现,Fusobacterium 的存在与免疫检查点阻断(Immune Checkpoint Blockade, ICB)治疗的反应在非小细胞肺癌(NSCLC)中呈负相关。具体来说,高Fusobacterium含量的肿瘤(Fuso-high tumors)显示出较低的细胞毒性、干扰素-γ(IFNG)和主要组织相容性复合体(Major Histocompatibility Complex, MHC)II类基因表达特征。这提示Fusobacterium可能通过干扰肿瘤内免疫环境来降低免疫治疗的有效性。
微生物对肿瘤微环境和宿主免疫反应的影响
微生物多样性与肿瘤免疫排斥和渗透
研究表明,微生物多样性与肿瘤微环境中的免疫细胞渗透有密切关联。具体来说,高微生物多样性与癌相关成纤维细胞(Cancer-Associated Fibroblasts, CAFs)的浸润和免疫排斥(immune exclusion)的表达特征呈正相关。这些发现表明,丰富的微生物群落可能通过促进CAF浸润和其他抑制性免疫环境改变来增强肿瘤的免疫排斥能力。
Toll样受体(Toll-Like Receptors, TLRs)介导的免疫调节
Toll样受体是一类能够识别微生物组成分并激活免疫反应的受体。研究显示,肿瘤微生物通过激活TLR4可诱导肿瘤微环境中的局部LPS/TLR4反应,从而模仿现有TLR激动剂疗法的效果。这一过程通过改变肿瘤微环境的动态和促进炎症反应,间接影响宿主的免疫反应。具体来说,TLR特异性疗法的开发是由于其作为免疫调节剂的能力,特别是在药物开发中越来越被重视。
微生物与细胞因子和信号通路的相互作用
微生物群落通过影响细胞因子和信号通路,如转化生长因子-β(Transforming Growth Factor-β, TGF-β)和肿瘤坏死因子-α(Tumor Necrosis Factor-α, TNF-α)的激活来调节免疫反应。这些细胞因子是由微生物衍生产品诱导的,它们在宿主-微生物相互作用中发挥核心作用,特别是在肠道。通过这些因子的激活,微生物可以影响肿瘤微环境的免疫特性,促进肿瘤的存活和扩散。此外,研究还观察到与基质金属蛋白酶(Matrix Metalloproteinases, MMPs)和肌动蛋白细胞骨架重塑因子的表达变化相关的细菌诱导的细胞外基质(Extracellular Matrix, ECM)改变。
潜在的局限性
样本来源的局限性:尽管研究涉及了多种癌症和多个器官的样本,但这些样本主要来源于一个特定的数据集,可能无法全面代表其他种族或地区的患者群体。
因果关系不明确:该研究强调了微生物多样性与肿瘤微环境(tumor microenvironment, TME)的相关性,但仅是观察性的关联,未能确立微生物与肿瘤特性之间的因果关系。
生物信息学方法的局限性:尽管使用了先进的生物信息学工具,如Kraken2和PathSeq,进行微生物组的分析,这些方法可能仍受限于技术误差和生物样本中微生物DNA含量的波动。
时间跨度和动态监测的不足:虽然研究考虑了治疗前后的微生物组变化,但缺乏更长时间跨度的动态监测,可能无法完全揭示治疗影响下的微生物组时间演变。
外部验证的缺失:该研究的发现主要基于内部数据集和分析,缺乏来自其他独立研究的外部验证,这可能限制了结果的普适性和验证性。
微生物定量的精确性:虽然使用了基于组装的宏基因组(assembled metagenomics)方法来提高微生物检测的精度,但在物种水平上的定量可能仍受限于当前的宏基因组技术和数据解析的复杂性。
潜在的研究方向
进一步探索肿瘤微生物组与免疫治疗效果的关联:研究已经指出肿瘤微生物组的多样性与免疫检查点阻断(immune checkpoint blockade, ICB)的反应存在相关性。未来可以通过更大规模的临床样本来验证微生物组成分与免疫治疗效果之间的关系,并探索是否可以通过调整微生物组成分来提高治疗效果。
微生物组在肿瘤微环境中的功能角色:研究表明,微生物能够影响肿瘤微环境中的炎症和免疫反应。未来的研究可以着重于解析特定微生物种类如何通过与宿主细胞的直接互作影响肿瘤的生长和扩散。例如,探索厌氧菌(anaerobic bacteria)在低氧(hypoxia)肿瘤微环境中的生存策略及其生物学效应。
微生物组和癌症治疗耐药性的关系:这项研究发现某些微生物如梭状杆菌属(Fusobacterium)与免疫治疗的反应差相关。未来可以探索其他类型的癌症和治疗方法中微生物组的影响,及其如何通过影响肿瘤细胞的药物代谢和肿瘤微环境来导致治疗耐药。
时间动态研究和微生物组变化:研究表明,在治疗前后微生物组的组成会发生变化。未来的研究可以通过长期追踪分析,观察治疗过程中微生物组如何变化,以及这些变化如何与疾病的进展和治疗响应相关。
跨癌种(cross-cancer)比较分析:通过比较不同类型癌症的微生物组,可以揭示哪些微生物群体是特定癌种或肿瘤微环境的特征,从而为癌症的分类和精准治疗提供新的生物标志物(biomarkers)。
原文链接
Battaglia TW, Mimpen IL, Traets JJH, van Hoeck A, Zeverijn LJ, Geurts BS, de Wit GF, Noë M, Hofland I, Vos JL, Cornelissen S, Alkemade M, Broeks A, Zuur CL, Cuppen E, Wessels L, van de Haar J, Voest E. A pan-cancer analysis of the microbiome in metastatic cancer. Cell. 2024 Apr 5:S0092-8674(24)00312-X. Epub ahead of print. PMID: 38599211.
DOI:https://doi.org/10.1016/j.cell.2024.03.021