当单个细胞发育成为一个完整有机体时,会产生巨大的细胞多样性。近年来,单细胞RNA测序(scRNA-seq)的高速发展使得无偏倚地定义反映个体发育、功能或解剖位置的细胞类型成为可能,但在完整的生物系统中对这些细胞进行高通量映射仍是一个技术挑战。开发能够在正常组织生理或肿瘤微环境背景下实现高度多重映射的技术,可为人类健康和疾病等多种生物学过程提供更多宝贵见解。
将空间索引与下一代测序技术相结合,可实现测序reads的空间映射和细胞类型的原位重建,但基于测序的空间转录组学方法受到RNA扩散和捕获效率的限制。细胞类型也可以通过多重单分子荧光原位杂交(smFISH)技术或原位测序靶向RNA来成像,该方法具有高度定量性并可扩展到整个转录组,但其需要高分辨率显微镜且过程繁琐;另一种方法是多重免疫染色或空间蛋白组学,其相比smFISH检测性能进一步提升,但成本较高、检测通量较低。
近日,新加坡基因组研究所的科研人员在Nature Communications上发表了题为“Highly sensitive spatial transcriptomics usingFISHnCHIPs of multiple co-expressed genes”的文章。研究团队提出了一种灵敏、稳健、可扩展的空间转录组学方法“FISHnCHIPs”,可对细胞类型和基因表达程序进行原位分析。FISHnCHIPs通过对组织中空间共定位的2-35个共表达基因(聚类成模块)同时成像来实现这一目标,从而获得与单基因FISH相似的空间信息,但灵敏度提高约2-20倍。利用FISHnCHIPs,研究团队对小鼠肾脏和小鼠大脑中多达53个模块进行了成像,并展示了对整个组织切片的高速、大视野的分析。此外,FISHnCHIPs还揭示了人类结肠癌活检中癌症相关成纤维细胞的空间限制定位。总体而言,FISHnCHIPs能够迅速对生理正常或正在发病的组织进行细胞分型。

文章发表在Nature Communications
共表达基因在组织内的相同细胞中存在空间共定位,因此可以设计靶向大量基因的RNA FISH探针,从而更可靠地检测任何感兴趣的细胞群。基于此,研究团队开发了FISHnCHIPs,其能在保留组织结构的同时准确地映射细胞类型。
为初步验证上述假设,研究团队使用小鼠肾脏scRNA-seq数据集设计了FISHnCHIPs探针,用于五种特定的细胞类型:肾巨噬细胞、肾小球内皮细胞、Henle细胞环(LOH)、集合管细胞(CD)和肾小球足细胞(图1)。研究团队观察到,每种细胞类型中前两个共表达基因之间存在高度共定位,证实了来自scRNA-seq中的相关基因在同一细胞中存在空间共定位。当使用14-23个基因的组合来标记这些细胞类型时,与仅标记单个最高差异表达(DE)基因相比,细胞更容易被检测到;FISHnCHIPs还揭示了肾脏组织结构的许多复杂细节。此外,与smFISH相比,在五种细胞类型中,每个细胞的FISHnCHIPs荧光强度增加约6-39倍。
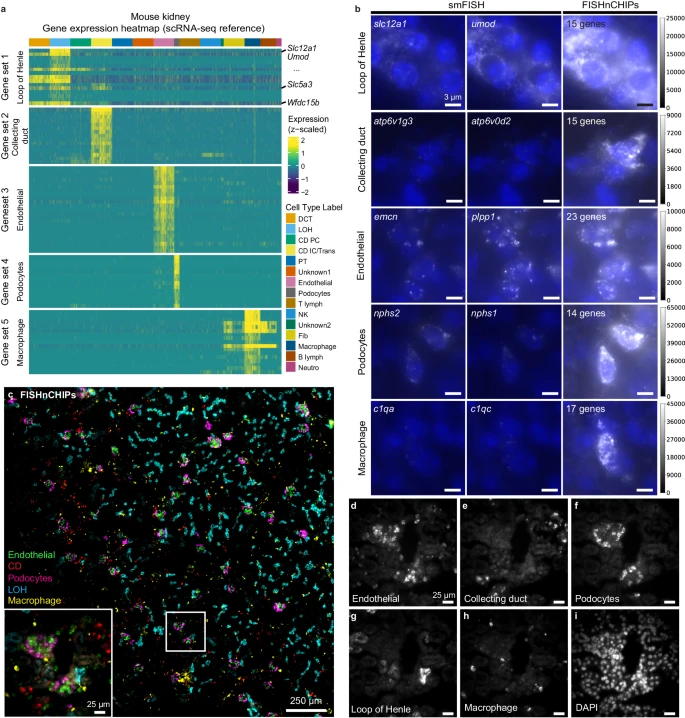
图1:小鼠肾脏组织中FISHnCHIPs和smFISH结果比较。
研究团队设计了基于基因模块的FISHnCHIPs检测方法,以自然减少串扰(图2):对小鼠视觉皮层数据集进行基因-基因相关矩阵的聚类,选择了255个与至少三个基因高度相关的候选基因,并确定了18个具有基因本体(GO)显著富集的基因模块。通过对基因模块进行分析,发现FISHnCHIPs信号的亮度比使用单个标记基因进行分析高1.2-22.3倍。
研究团队还使用自动流控系统对新鲜冷冻小鼠脑组织切片中的每个基因模块依次成像,并创建了一个软件框架,根据FISHnCHIPs数据对细胞类型进行对齐、分割和聚类。对来自6,180个细胞的基因模块无监督、基于图的聚类,并再现了主要脑细胞类型的预期频率和空间分布,包括谷氨酸能神经元、GABA能神经元、星形胶质细胞等。FISHnCHIPs检测到的细胞类型频率与MERFISH高度相关,并且在不同技术重复中都能可靠地检测到。
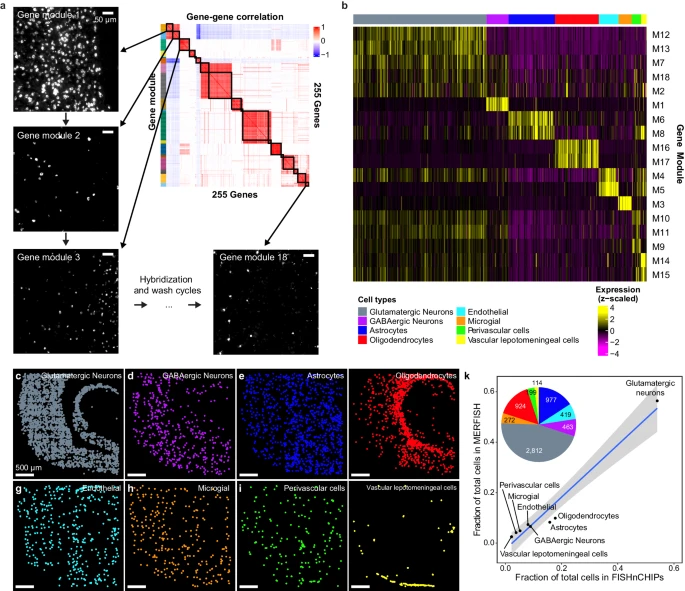
图2:在小鼠皮层中对18个基因模块进行FISHnCHIPs分析。
接下来,研究团队展示了FISHnCHIPs可用于区分视觉皮层典型层结构的神经元亚型;对先前注释的20个特征和活性基因表达程序进行了基因-基因相关分析,并设计了一个FISHnCHIPs panel,每个程序平均包含16个基因(图3)。结果显示,通过FISHnCHIPs可以更清晰地检测神经元程序:14个特征程序(ExcL2、ExcL3... Sub)在空间上更局域化,6个活性程序(Erp、LrpD... Syn)在空间上表达更普遍。特别地,FISHnChIPs还揭示了这些基因表达程序之间微小的空间变化。
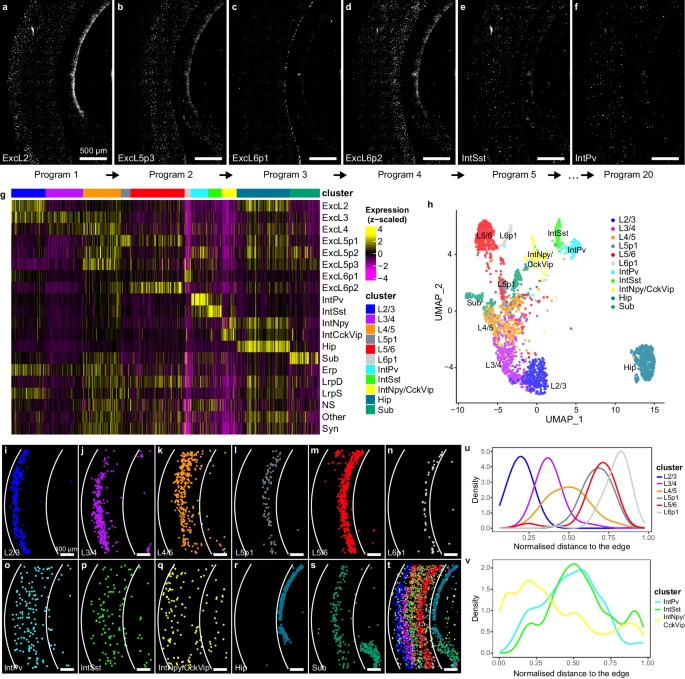
图3:在小鼠皮层中对20个基因表达程序进行FISHnCHIPs分析。
除分析模块和程序的可靠性外,与传统基于单基因的方法相比,FISHnCHIPs能够提高成像通量,在相同的时间内实现更大的视野和更多的细胞分析。研究团队利用未排序的scRNA-seq数据,设计了包含53个模块(674个基因)的FISHnCHIPs检测panel,以对小鼠大脑中神经元和非神经元细胞进行更全面的细胞分型。结果显示,53个模块panel的ARI得分为0.814,表明其可以在很大程度上重现已知的脑细胞类型。
为进一步评估高通量FISHnChIPs方法的检测性能,研究团队使用两个紧密相邻的冷冻切片,比较了在10×和60×物镜下观察到的细胞类型的频率和空间分布。结果显示,10×和60×数据集之间的聚类大小高度相关,表明通量增加的同时数据质量没有降低。此外,与先前文库相比,53模块文库中的细胞通过质量控制(QC)的比例较高(97%),表明当FISHnChIPs被设计用于针对广泛的细胞类型,几乎所有成像的细胞都能得到分析,进一步证明了FISHnChIPs的高检测效率和灵敏性。
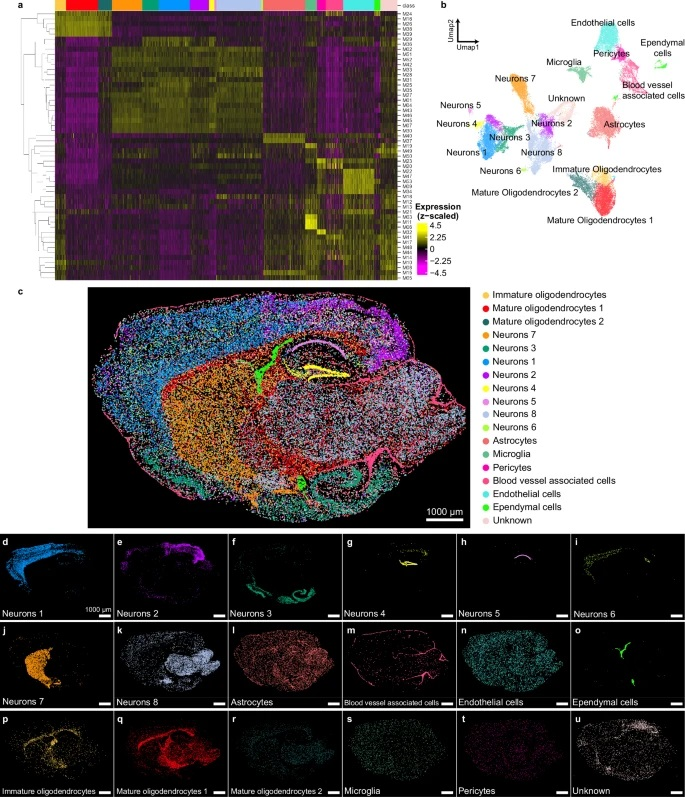
图4:小鼠大脑中53个基因模块的大FOV FISHnCHIPs分析。
最后,研究团队展示了FISHnCHIPs的稳健性,利用其分析了人类结肠癌(CRC)组织的冷冻活检样本,通过成像分析了两种已鉴定的癌症相关成纤维细胞(CAF)亚型,并在CRC组织中共染了上皮细胞(肿瘤标记基因)和免疫细胞(HLA基因)(图5)。结果显示,两种CAF亚型具有不同空间组织,其中表达肌肉收缩程序的CAF-2亚型似乎促进了免疫抑制微环境的形成。
此外,FISHnCHIPs的染色模式与免疫荧光(IF)标记一致,相比之下,针对单基因DCN/MMP2(CAF-1标记物)以及TAGLN/ACTA2(CAF-2标记物)的smFISH结果较暗淡,这凸显了使用多个共表达基因标记细胞类型的益处。上述结果表明,FISHnCHIPs可稳健地成像小鼠和人类组织中的细胞类型,以及通过无监督聚类来表征细胞类型。
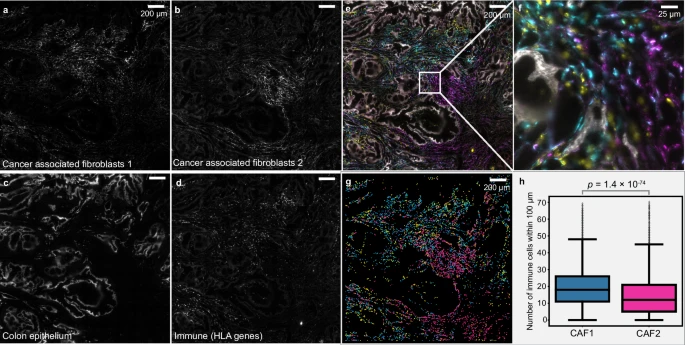
图5:FISHnCHIPs成像CRC组织中癌相关CAFs亚型。
综上所述,研究团队开发了一种基于基因模块的 FISHnCHIPs检测方法,可通过多个共表达基因来分析单细胞,并利用相关基因的冗余性来提高检测灵敏度和稳健性。与传统的FISH相比,FISHnChIPs的细胞类型映射灵敏度提高约2-20倍。总之,FISHnChIPs的高灵敏度和大规模分析能力有助于人们理解复杂组织和细胞群体,特别是在识别罕见细胞类型和揭示微环境内细胞间相互作用的复杂性方面。
参考文献:
Zhou, X., Seow, W.Y., Ha, N. et al. Highly sensitive spatial transcriptomics using FISHnCHIPs of multiple co-expressed genes. Nat Commun 15, 2342 (2024).
https://doi.org/10.1038/s41467-024-46669-y