腺嘌呤单碱基编辑器(ABE)是一种可以精确高效地将腺嘌呤转化为鸟嘌呤的基因编辑工具,有潜力用于治疗单核苷酸变异(SNV)造成的遗传疾病。SNV是造成遗传型心肌病的主要原因之一。大量心肌病致病突变有可能被ABE纠正。过去三年间,基于ABE技术在小鼠和人源心肌模型中纠正心肌病致病突变的工作被大量报道[1-4],展现出ABE技术颠覆心脏基因疗法的巨大潜力。然而,针对心脏的ABE载体的设计和构筑方法尚未成熟,其中关键的技术难点和潜在问题尚需进一步解决。
2024年2月8日,北京大学基础医学院、北京大学心血管研究所、血管稳态与重构全国重点实验室郭宇轩研究员团队在Journal of Cellular and Molecular Medicine在线发表了题为“Adenine base editor-based correction of the cardiac pathogenic Lmnac.1621C > T mutation in murine hearts”的文章[5]。该研究发现:①DeepABE[6]是有效的评估心脏ABE编辑效果的预测软件;②基于降低PAM限制的ABE变体,在靶位点附近筛选sgRNA,有可能降低旁观者效应,提高ABE的精确性;③改变ABE中TadA8e和Cas9变体的组合有助于提高ABE编辑效率;④利用紧凑型Cas9变体[7]可以实现单个AAV递送的心脏ABE编辑。

1. DeepABE预测工具在小鼠心脏基因编辑中的应用
ABE对于靶基因位点的有效编辑很大程度上依赖于sgRNA的设计与筛选,以往研究中对于sgRNA的筛选主要基于在干细胞或动物模型中检测既定的腺嘌呤的编辑效率的方式,实验周期长且花费成本较高,为了解决这一问题,研究人员利用一种基于计算机的深度学习模型—DeepABE来预测ABE在小鼠心脏中的碱基编辑效率。
首先,研究人员针对已发表的四项代表性的使用ABE治疗小鼠心肌病研究中的数据,利用DeepABE对于ABE的编辑效率进行预测,并与已发表的数据进行相关性分析。结果显示这两者具有高度相关性,表明DeepABE能有效预测ABE在小鼠体内的编辑效率(Figure 1A-B)。接着,研究人员针对新的扩张型心肌病小鼠模型[8] Lmnac.1621C>T致病突变位点设计了5条sgRNA,其中c.1621T位点为靶编辑位点,c.1613T和c.1619T为潜在的旁观者效应(bystander effect)位点。过高的旁观者效应产生可能会导致难以预测的严重后果,因此,研究人员利用DeepABE筛选了高靶基因位点编辑效率且低旁观者效应的3条sgRNA (sgRNA1-3)以进行下一步实验(Figure 1C-D)。
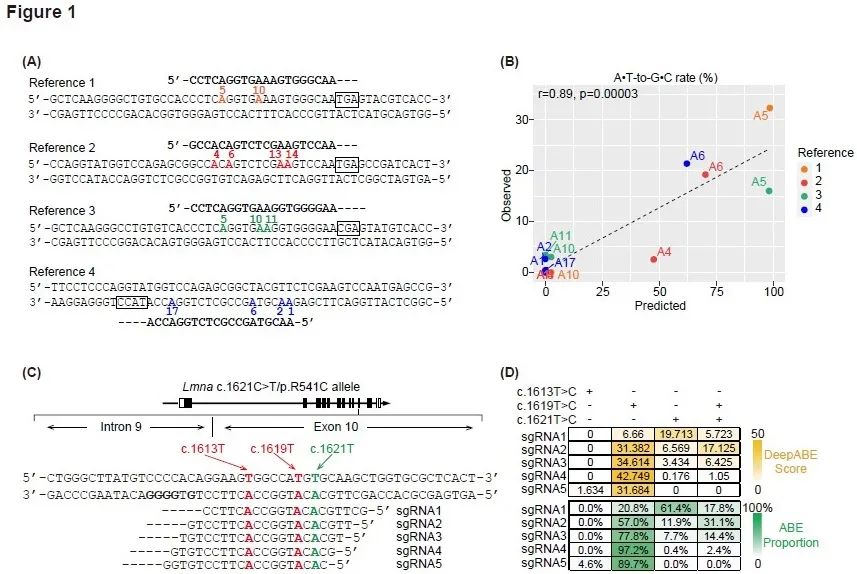
为了评估DeepABE预测的准确性,研究人员利用双AAV9-ABE载体对Lmna c.1621C > T突变进行纠正。双AAV-ABE系统是利用Split-intein技术,将ABE基因拆成两半,分别由两个不同的AAV载体进行基因递送,当这两个AAV载体进入同一个细胞,同时表达两种蛋白片段时,Intein介导蛋白融合形成全长的TadA-nSpCas9蛋白,从而进行ABE编辑(Figure 1E)。对携带Lmna c.1621C>T突变的新生小鼠(LmnaRC/RC小鼠)进行单次皮下注射双AAV9-ABE载体后,扩增子测序发现sgRNA1能在心脏和肝脏组织中同时实现最高的靶位点的突变纠正效率和最低的旁观者效应,同时DeepABE预测结果与实验结果具有很强的相关性,进一步证实了DeepABE可作为ABE在心脏基因编辑中的预测工具(Figure 1F-G)。
2. 改进ABE元件提高心脏基因编辑效率
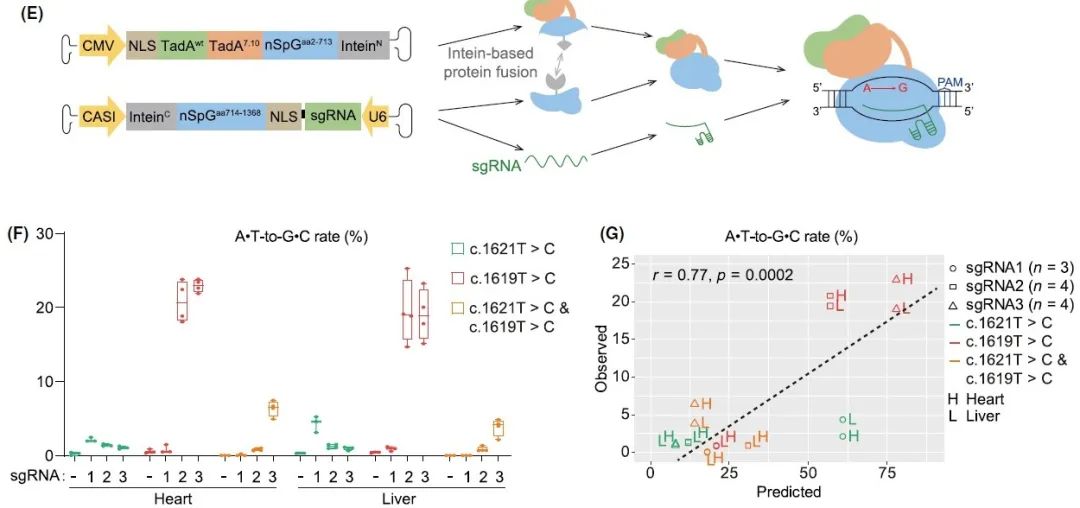
由于在利用TadA7.10-SpG系统(Figure 1E所示)纠正Lmnac.1621C>T突变过程中,研究人员发现这一系统对于纠正心脏中突变位点的效率较低(<5%)。因此,研究人员试图通过改造ABE元件,更换TadA和Cas9,以提高心脏组织中的基因编辑效率。
TadA自然条件下以同源二聚体形式存在,在ABE7.10[9]系统中,由一个野生型的TadA融合一个工程化的TadA7.10突变体形成二聚体以增强ABE的活性,而在后续的研究中,TadA7.10进化为TadA8e,ABE8e[10]系统中不再需要两个TadA融合就能充分发挥其性能,因此,研究人员首先基于sgRNA1和SpG对比了ABE7.10和ABE8e系统,对LmnaRC/RC新生小鼠注射等剂量的AAV-ABE7.10和AAV-ABE8e病毒,扩增子测序结果显示,ABE8e在心脏和肝脏组织中均能检测到致病突变的成功纠正,同时心脏组织中能提高4.9倍的靶基因位点(Lmna c.1621T)的编辑效率,同时不影响c.1619位点的编辑(Figure 2A-B)。
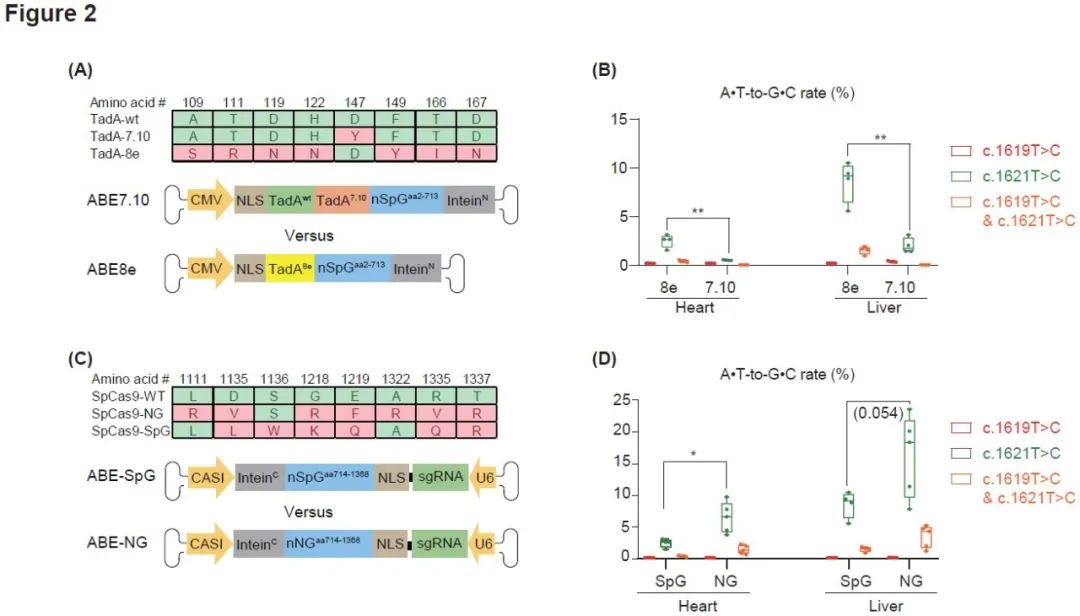
接着,研究人员试图通过改变Cas9同系物来提高ABE的编辑效率。Cas9存在不同的工程化改造后的变体。SpG和NG是两个独立进化但具有相同的简化的PAM序列(NGN)的SpCas9突变体,两者之间仅有7个氨基酸序列不同,且这7个氨基酸都位于Cas9 C端的AAV载体中,因此研究人员构建了TadA8e-SpG和TadA8e-NG两种载体,递送相同剂量AAV病毒至LmnaRC/RC新生小鼠体内,结果显示,两种系统均能实现对突变位点的成功纠正,基于NG的AAV-ABE系统对于心脏组织中Lmnac.1621C>T位点的纠正效率比SpG提高约2.5倍(Figure 2C-D)。这些结果表明,优化TadA、Cas9等ABE的核心元件能有效提高心脏中的碱基编辑效率。
3. 实现单AAV-ABE载体的体内基因递送
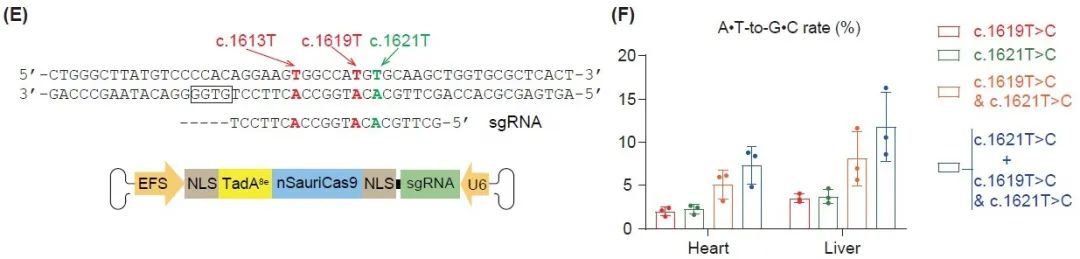
双AAV系统存在所需病毒剂量大,潜在副作用大,病毒包装成本高及载体设计复杂等问题。最后,研究人员通过改变Cas9同系物及启动子,将SpCas9突变体替换为小型的SauriCas9突变体,将CMV启动子替换为小型的EFS启动子,实现单AAV递送的ABE载体。由于SauriCas9的PAM序列为NNGG,与SpCas9 (NGG)十分相似,因此研究人员可沿用sgRNA1的序列设计,重新组装了单AAV-ABE载体。研究人员注射双AAV-ABE载体剂量一半的单AAV载体至LmnaRC/RC新生小鼠体内,在心脏和肝脏组织中均能检测到Lmnac.1621C>T突变位点的纠正(Figure 2E-F)。但是由于改变了Cas9的性质,sgRNA1原有的降低旁观者效应的能力消失了。单AAV-SauriABE载体处理的心脏中,多数被编辑的基因组发生了Lmnac.1621T+c.1619T的联合碱基编辑。
总体而言,本研究证实了DeepABE对体内基因编辑效率的预测作用,但是这种作用可能仅适用于SpCas9来源的ABE。本研究构建了能在体内纠正Lmnac.1621C>T突变的AAV-ABE载体。优化AAV和ABE可以提高体内基因编辑效率,但是目前本载体的编辑效率仍旧有限,不足以实现心脏表型的逆转。另外,本研究初步实现了单AAV递送ABE对心脏的碱基编辑作用,为进一步简化AAV-ABE载体设计打下了基础。
北京大学心血管研究所郭宇轩研究员和首都医科大学附属安贞医院高霏教授是本研究的共同通讯作者。北京大学博士研究生杨璐梓和刘占钊、科研助理孙金焕和本博联读生陈展是共同第一作者。本研究获得国家自然科学基金、国家重点研发计划和北京市科技新星计划的资助。
参考文献:
[1] Koblan, Erdos, Wilson, Cabral, Levy, Xiong, Tavarez, Davison, Gete, Mao, Newby, Doherty, Narisu, Sheng, Krilow, Lin, Gordon, Cao, Collins, Brown, Liu. In vivo base editing rescues Hutchinson–Gilford progeria syndrome in mice. Nature, 2021, 589(7843): 608-14.
[2] Grosch, Schraft, Chan, Küchenhoff, Rapti, Ferreira, Kornienko, Li, Radke, Krämer, Clauder-Münster, Perlas, Backs, Gotthardt, Dieterich, Van Den Hoogenhof, Grimm, Steinmetz. Striated muscle-specific base editing enables correction of mutations causing dilated cardiomyopathy. Nature Communications, 2023, 14(1):
[3] Chai, Cui, Chemello, Li, Chen, Tan, Atmanli, Mcanally, Zhang, Xu, Liu, Bassel-Duby, Olson. Base editing correction of hypertrophic cardiomyopathy in human cardiomyocytes and humanized mice. Nature Medicine, 2023, 29(2): 401-11.
[4] Reichart, Newby, Wakimoto, Lun, Gorham, Curran, Raguram, Delaughter, Conner, Marsiglia, Kohli, Chmatal, Page, Zabaleta, Vandenberghe, Liu, Seidman, Seidman. Efficient in vivo genome editing prevents hypertrophic cardiomyopathy in mice. Nature Medicine, 2023, 29(2): 412-21.
[5] Yang, Liu, Sun, Chen, Gao, Guo. Adenine base editor-based correction of the cardiac pathogenic Lmna c.1621C > T mutation in murine hearts. J Cell Mol Med, 2024, 28(4): e18145.
[6] Song, Kim, Lee, Kim, Seo, Park, Choi, Jang, Shin, Min, Quan, Kim, Kang, Yoon, Kim. Sequence-specific prediction of the efficiencies of adenine and cytosine base editors. Nat Biotechnol, 2020, 38(9): 1037-43.
[7] Davis, Wang, Witte, Huang, Levy, Raguram, Banskota, Seidah, Musunuru, Liu. Efficient in vivo base editing via single adeno-associated viruses with size-optimized genomes encoding compact adenine base editors. Nat Biomed Eng, 2022, 6(11): 1272-83.
[8] Yang, Sun, Chen, Liu, Sun, Lin, Hu, Zhao, Ma, Lu, Li, Guo, Dong. The LMNA p.R541C mutation causes dilated cardiomyopathy in human and mice. Int J Cardiol, 2022, 363(149-58.
[9] Gaudelli, Komor, Rees, Packer, Badran, Bryson, Liu. Programmable base editing of A•T to G•C in genomic DNA without DNA cleavage. Nature, 2017, 551(7681): 464-71.
[10] Richter, Zhao, Eton, Lapinaite, Newby, Thuronyi, Wilson, Koblan, Zeng, Bauer, Doudna, Liu. Phage-assisted evolution of an adenine base editor with improved Cas domain compatibility and activity. Nat Biotechnol, 2020, 38(7): 883-91.