【衡道丨笔记】NGS检测原理
时间:2024-03-09 16:03:42 来源 网络 作者:网络
内容摘要
一、NGS的发展与主流平台介绍
二代测序的应用领域
-
无创产前检测
-
遗传性肿瘤诊疗
-
肿瘤靶向诊疗
-
病原微生物检测
-
测序技术的迭代发展
二代测序技术主流平台

二代测序的常见术语
Reads数:
测序时每产生一条序列称为一个Read,一次测序完成后产生的所有序列总数即为Reads数。
测序读长:
测序时一次反应所读取的序列长度,如2x150 bp即表示双端各读取150bp长度的序列。DNA文库的插入片段长度必须在读长范围内。
测序通量:
一次测序所产生的数据总量,可理解为(Reads数x读长)。
测序深度:
目标基因组中特定核苷酸被测的次数,可理解为测序得到的数据总量与目标基因组大小的比值。
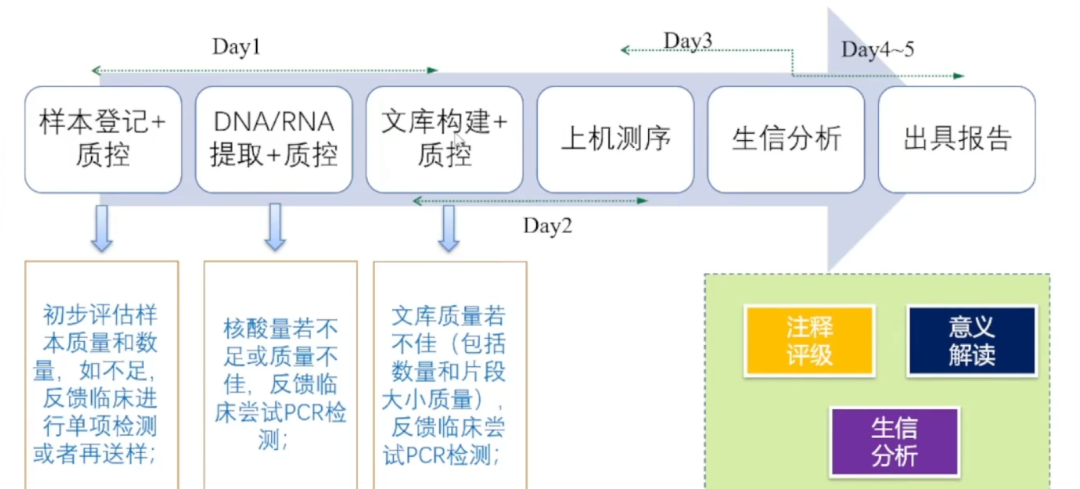
一、NGS建库测序原理及实验流程
建库三大平台
1、扩增子平台
-
针对性强
-
可达成千上万重扩增反应同时进行
-
操作快速简便
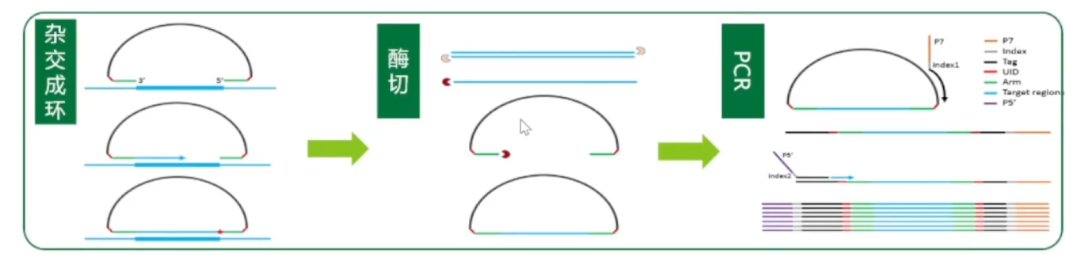
2、杂交捕获平台
-
目标区域容量大,通用性强
-
可添加UID,灵敏度更高
-
满足ctDNA检测需求
-
检测流程长,考虑报告周期
流程:
预文库→杂交→捕获→洗涤→扩增捕获文库
3、HANDLE平台
-
极简操作,单管完成,<1h手工操作
-
可添加UID,特异性出众
-
多种变异类型同时检测
-
适合临床检测
流程:
主流平台测序原理
1、IlIumina平台测序原理:
碱基互补配对原则:
1、在DNA聚合酶的作用下连接一个带荧光的碱基
2、清除未反应的碱基和酶
3、激发荧光信号并读取
4、去除荧光基团和阻断基团
2、lon Torrent平台测序原理:
乳液PCR:做好的文库,种到测序珠子上进行扩增。
3、ADx-SEQ200Plus测序原理:
DNA纳米球→阵列式流动槽→联合探针锚定聚合技术
三、NGS基于HGVS的基因变异命名规范
-
所有变异类型的描述应该基于DNA水平上的改变,RNA或蛋白质水平的改变可以作为补充。
-
确认所描述的变异是实验所得还是理论推断所得,如果是理论推断所得要加括号。
-
RNA或蛋白质水平的检测结果的描述不能用于反推DNA的改变(描述),因为转录/翻译方式、简并密码子等问题。
-
使用公开且描述清晰的参考序列。
-
参考序列使用NCBI或EBI数据库中的ID,必须同时包含accession和version信息。
-
如果在NCBI和EBI中没有合适的参考序列,可申请一个LRG编号。
-
标记变异所在位置时使用参考序列的首字母缩写。
-
变异位置与参考序列文件标识符之间用“:”隔开,例如 NM 000059.3:exon23:c.9085G>A:p.(A3029T)。
-
基因组序列(前缀为9.):位置1指第一个基因组第1个核苷酸(不可使用+、-或其他符号标志)。
-
编码DNA(前缀为c.):位置1指启动密码子ATG中的第一个核苷酸A。
-
RNA(前缀为r.):与编码DNA类似。
-
蛋白质(前缀为p.):位置1指蛋白质第一个氨基酸。
-
也适用于单碱基或随机串联重复(氨基酸和核苷酸)。
-
所有水平的描述均适用(genome,gene,transcript,protein)
-
特例:使用c.,r. or n.描述外显子/外显子连接区域的缺失/重复时不适用。
-
DNA-level 123456A>T。
-
RNA-level 76a>u。
-
protein level Lys76Asn(三字母优先于单字母)。
-
国际生化联合命名委员会(NC-IUB)的命名规范。
-
当一个变异同时适用重复或插入的变异类型时,根据优先级应描述为重复。
-
缺失一部分序列不可再用一部分相同的序列替代。
-
间隔一个核苷酸的两个变异,如果影响同一个氨基酸,应描述为“delins”。
-
新版的规则将改为:两个变异被少于两个核苷酸隔开时应描述为“delins“。
NGS在临床中的应用
基于扩增子建库的Classic Panel:
适合院内落地的靶向用药NGS检测首选
-
一次性检测包括肺癌8个核心基因、NTRK1/2/3等在内的40个靶向用药基因。
-
涵盖肺癌、肠癌、胃癌、食管癌、甲状腺癌等20+癌种;涉及已获批或正在临床试验中的70+药物。
-
涵盖部分免疫治疗相关基因。
基于HANDLE建库的Classic Panel NGS产品:
简单快速,更易于院内开展