近年来,单分子荧光原位杂交(smFISH)方法的出现使人们能够同时检测单细胞中的几种RNA分子。鉴于其高灵敏度,smFISH已成为检测RNA原位表达的金标准,并被用于揭示RNA定位在细胞迁移、神经元连接和局部蛋白质合成中的重要性。但smFISH受到荧光团光谱重叠的限制,其复用能力有限,在解析复杂组织中的细胞异质性等任务方面扩展性不佳。
目前,可组合编码的原位杂交技术可利用数十个FISH循环解码数百甚至数千种RNA的特性。与smFISH相比,这些方法的多重性增加了2-3个数量级,但其需要更长的靶RNA转录本(>1.5kb),限制了对重要分子的分析。此外,基于测序的空间转录组学方法的出现,帮助人们实现了组织切片的转录组全谱分析。但与原位方法相比,基于测序的空间转录组学方法通常具有较低的捕获效率、复杂的载玻片制备程序以及较高的测序成本等。
加州大学圣地亚哥分校张鹍教授团队在Nature Communications发表了题为“Mapping human tissues with highly multiplexed RNA in situ hybridization”的文章。研究团队介绍了最新开发的DART-FISH技术(Decoding Amplified taRgeted Transcripts with Fluorescence in situ Hybridization),能够在厘米大小的人体组织切片中分析数百至数千个基因。DART-FISH主要技术特点包括:稳健的条形码方案、一套用于在大型池中生产锁式探针(padlock probe)的分子方案、原位挂锁(padlock)捕获和扩增、一种名为RiboSoma的细胞质染色、等温和无酶解码,以及一种基于稀疏反卷积的计算方法,用于从密集荧光图像中解码像素级特征。
通过基准测试并与现有的方法进行比较,研究团队证明了DART-FISH的敏感性和特异性,重现了主要神经元和非神经元细胞类型的空间组织。此外,研究团队还使用DART-FISH技术检测了人体病变肾脏切片中的300个基因,表征了正常和疾病状态下细胞类型的空间分布。总体而言,DART-FISH工作流程为解决关键的生物学问题提供了新的方案,同时易于实施,不需要专门或定制的设备。

文章发表在Nature Communications
主要研究内容
DART-FISH整体工作流程
DART-FISH是通过锁式探针捕获目标转录本和滚环扩增(RCA)来产生原位特征,然后通过序列等温杂交进行高度稳健的解码。为评估人体组织中的RNA含量以及cDNA分子的原位保留情况,研究团队在反转录引物上添加了一个5’handle,以使所有具有荧光寡核苷酸的cDNA分子可视化,并将这一方法命名为“RiboSoma”。此外,为实现高复用性,研究团队使用组合标记来生成基因特异性条形码,使其可以在不到10轮成像的时间内产生足够的多样性,以编码数百到数千个基因。
值得注意的是,上述过程能够实现快速和可靠的解码,是因为其完全依赖于短寡核苷酸在室温下的杂交,无需复杂的温度控制装置,并避免了在显微镜下进行酶促反应的复杂性。因此,DART-FISH使用一种无酶和等温的方法来解码克隆,从而缩短了周期之间的准备时间。
最后,研究团队还开发了稀疏反卷积(SpD)解码算法,能够将反卷积形式化为正则化线性回归问题,其中条形码可以通过线性组合来形成观察到的像素强度,并在促进稀疏性的条件下优化组合,最终获得每个条形码的初始权重图。该计算框架可通过使用20倍物镜成像来减轻光学过度拥挤,并提高通量。

图1. DART-FISH工作流程。
DART-FISH的基准测试和验证
为评估DART-FISH对大型人体组织切片中超过100种RNA进行快速图像采集的性能,研究团队将其应用于新鲜冷冻人类初级运动皮层(M1C)脑切片中。
研究团队设计了5,097个探针,以选定的、包含已知标记基因的121个基因为靶点,解析了兴奋性和抑制性神经元以及非神经元细胞的空间组织。经图像预处理和SpD解码,共得到2,008,260份转录本;上述121个基因的表达水平在两个重复之间高度一致,表明DART-FISH具有较高的重复性。研究团队使用RiboSoma对细胞进行分割,发现更高比例的MBP转录本位于细胞外,反映了MBP转录本在轴突-胶质细胞接触位点的局部翻译。
为评估转录本定位的空间特异性,研究团队检测了兴奋性神经元中的标记基因SLC17A7和SATB2以及抑制性神经元中的GAD1和GAD2,并将10个标记基因的表达与RNAscope在平行M1C组织切片上生成的结果进行比较,发现RNAscope和DART-FISH具有高度一致性,表明DART-FISH可特异性地绘制这些标记基因在人类M1C中的空间定位。
以上结果表明,DART-FISH是一种可重复的空间转录组学方法,具有灵敏度和特异性,可在其空间环境中检测数百种RNA。

图2. 人类M1C脑切片上对DART-FISH进行基准测试和验证。
人类M1C细胞类型的组织结构
为验证DART-FISH能否解析人类M1C各种细胞类型的组织,研究团队对DART-FISH细胞进行聚类,并将其与M1C单核RNA测序(snRNA-seq)参考文献中相关性最高的亚类进行匹配。结果显示,从主要兴奋性、抑制性和非神经元细胞类别中共检测出20个亚类,分别在M1C中占24.3%、10.6%和65.1%。DART-FISH成功检测到大脑皮层中兴奋性神经元的层状组织和更广泛的抑制性神经元的层特异性;对于非神经元细胞,DART-FISH观察到少突胶质细胞出现在第4层,并在白质中达到顶峰。
此外,研究团队还利用DART-FISH检测到了短神经肽基因(如SST和NPY),以及其他短RNA物种,包括PCP4(534nt)和TMSB10(461nt),并验证了一个深层神经元标志物(TMSB10)。以上结果表明,DART-FISH可以准确地绘制人类大脑中所有主要的神经元和非神经元亚类的分布,并可通过检测短基因发现罕见的细胞群。
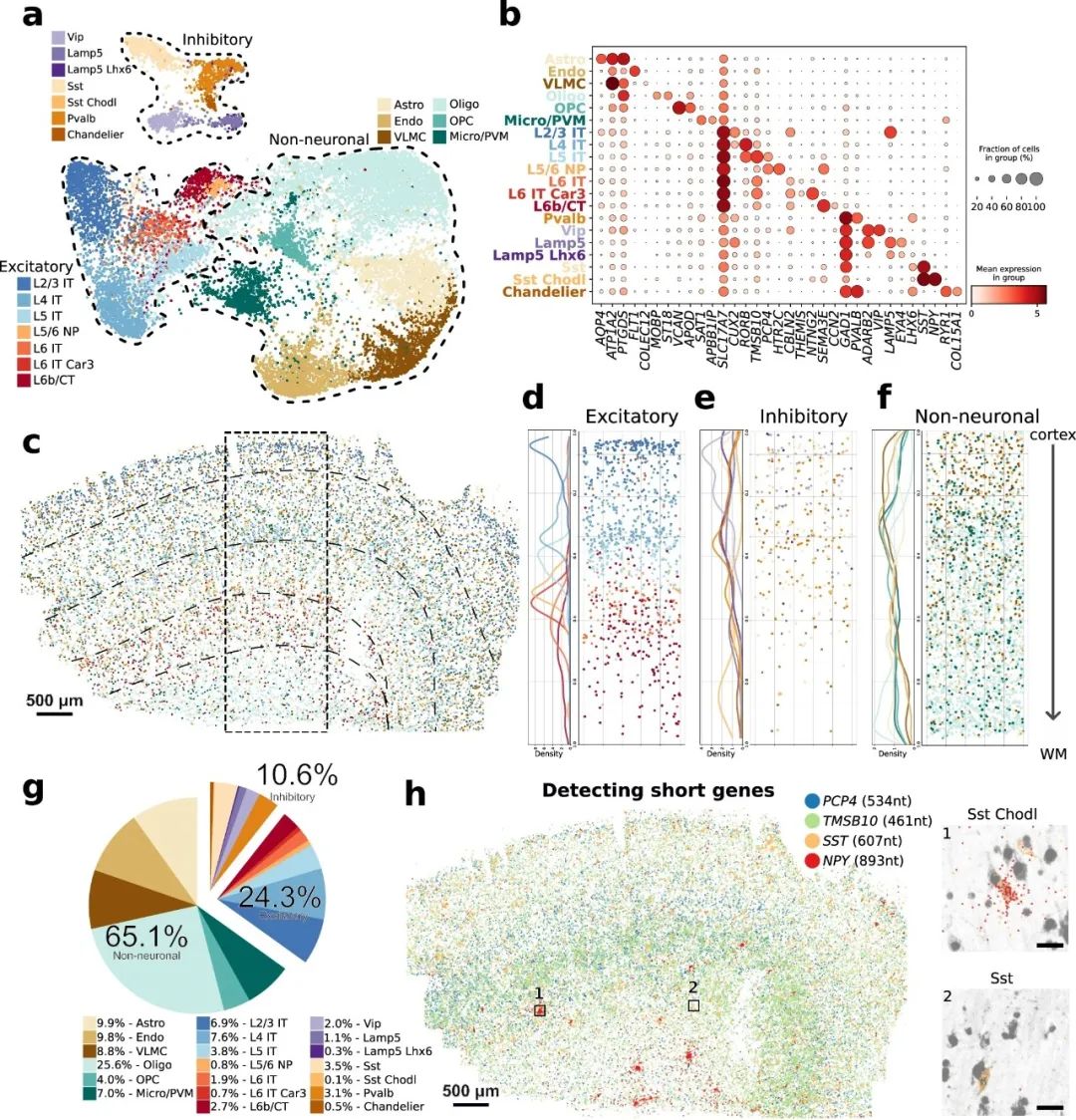
图3. 人类M1C细胞类型的DART-FISH图谱。
DART-FISH在异常人类肾脏中的应用
为证明在临床相关组织环境中的适用性,研究团队设计了一个包含300个基因的6,299个锁式探针,并对一个具有典型临床特征患者的肾皮质组织切片进行DART-FISH分析。结果显示,DART-FISH正确地绘制了肾单元不同区域的细胞空间组织,包括肾小球和皮质小管。此外,为确定人肾细胞的分子特性,研究团队使用RiboSoma和核染色进行细胞分割,发现RiboSoma在显示肾小管形态和区分间质细胞方面优于核染色。因此,DART-FISH可以准确分辨出人类肾脏中超20种细胞类型和状态。
接下来,研究团队分析了由健康细胞类型形成的邻域,并成功再现了肾小体、足细胞等人类肾脏内的已知复杂典型结构。除健康细胞类型外,DART-FISH还能够显示不同的病理细胞状态,包括表达基质基因的肌成纤维细胞(MYOF)(如COL1A1、TNC、DCN和POSTN),其分子分辨率超过传统组织学。基于DART-FISH的细胞类型注释与人类肾脏内已知的结构密切匹配,并且作为单细胞分辨率空间转录组学技术,DART-FISH可以用于探索患病人体组织中单细胞转录组测序研究所定义的细胞类型和状态。
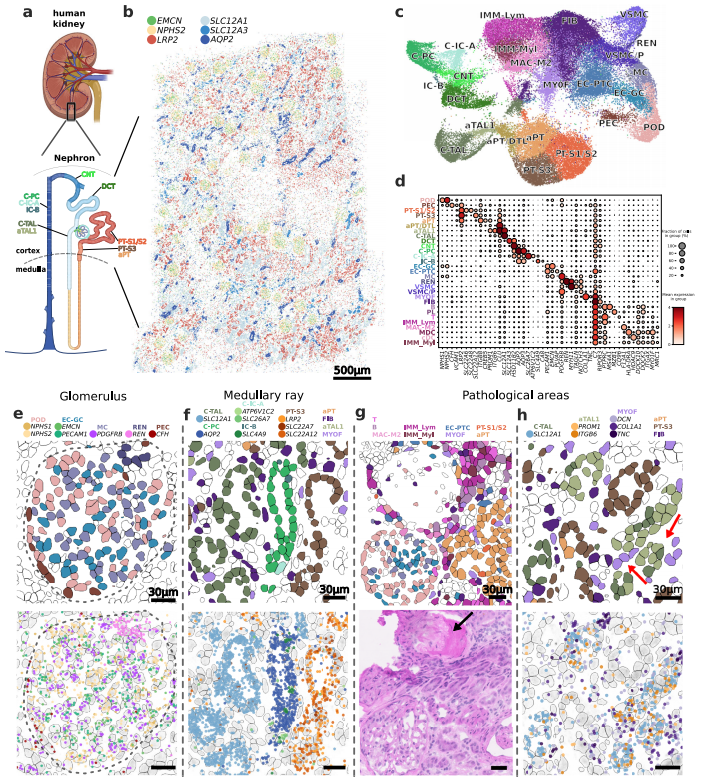
图4. DART-FISH在人类肾脏中的应用。
结语
综上所述,张鹍教授团队开发的DART-FISH是一种高通量RNA原位定位技术。在人脑中,DART-FISH重现了20种神经元和非神经元亚类的细胞结构;在肾脏中,DART-FISH解析了20个健康和病理细胞状态,并确定了富集在转录改变的上皮细胞和肌成纤维细胞中的病变微环境。DART-FISH是一种成本效益高的技术,无需特殊的设备,并且能够在相对较大的组织切片上应用,可助力科学家们对空间组织的研究。
论文原文:
Kalhor, K., Chen, CJ., Lee, H.S. et al. Mapping human tissues with highly multiplexed RNA in situ hybridization. Nat Commun 15, 2511 (2024). https://doi.org/10.1038/s41467-024-46437-y